COSMIC, der Catalogue Of Somatic Mutations In Cancer (Katalog der somatischen Mutationen bei Krebs), der vom Wellcome Trust Sanger Institute verwaltet wird, ist eine der größten und umfassendsten Ressourcen zur Erforschung der Auswirkungen somatischer Mutationen bei menschlichem Krebs. Sie ist das Ergebnis einer umfangreichen Datenkuratierung durch Experten und enthält Daten aus Tausenden von Veröffentlichungen und vielen wichtigen großen Krebsgenomik-Datensätzen. COSMIC dient als Hauptportal für mindestens vier wichtige Projekte oder Ressourcen: COSMIC – der Catalogue Of Somatic Mutations In Cancer, der Cancer Gene Census, das Cell Lines Project und COSMIC-3D. Akademische Nutzer können die Daten kostenlos nutzen und herunterladen (mit Registrierung), solange sie die Daten nicht weiter verbreiten und den Lizenzbedingungen zustimmen. Gewerbliche Nutzer müssen für das Herunterladen von COSMIC eine Gebühr entrichten. Diese Ressourcen bieten Zugang zu einer Vielzahl von Daten, Visualisierungen und Interpretationen zum Verständnis von Krebsmutationen und Krebsgenen.
Die COSMIC-Datenbank
Um COSMIC zu durchsuchen, können Sie einfach zur Hauptseite navigieren und im Suchfeld nach einem Gen, einer Krebsart, einer Mutation usw. suchen. Zur Veranschaulichung werden wir die Ergebnisse für ein einzelnes Gen untersuchen. Geben Sie BRAF in die Suchmaske ein und drücken Sie die Eingabetaste. Zum Zeitpunkt der Erstellung dieses Dokuments lieferte dieser Suchbegriff Ergebnisse für 1 Gen, 826 Mutationen, 49 Krebsarten, 226 Proben, 1575 Pubmed-Zitate und 0 Studien (siehe Ergebnisregisterkarten). Beachten Sie, dass COSMIC Ergebnisse für fast 300.000 getestete Proben und über 50.000 Mutationen in BRAF enthält.
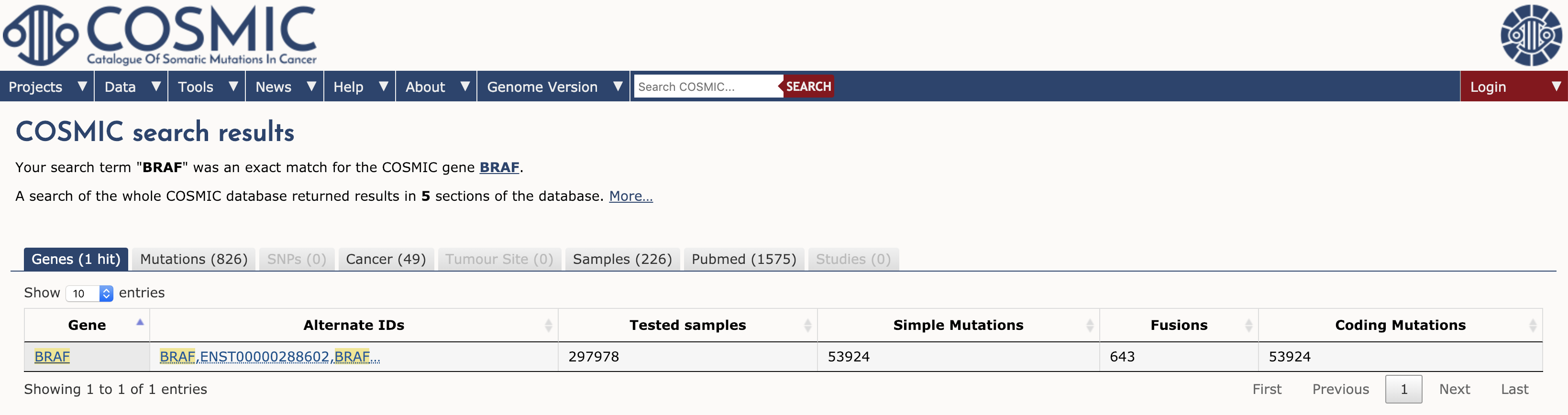
Wenn wir das BRAF-Ergebnis auswählen, gibt COSMIC eine detaillierte Seite zurück, die Folgendes bietet: Gen-Zusammenfassungen, Links zu anderen COSMIC-Ressourcen (z. B. Census-Gene, Hallmark-Gene usw.), externe Links, Arzneimittelresistenz, Gewebeverteilung, Genombrowser-Ansicht, Mutationsverteilung, Varianten und Referenzen. Wir werden uns einige dieser Abschnitte ansehen. Betrachten wir zunächst den Abschnitt Übersicht. Am oberen Rand dieses Abschnitts befinden sich mehrere nützliche Symbole. Das Symbol „Census-Gen“ zeigt an, dass BRAF ein bekanntes Krebsgen gemäß dem Gene Census ist (siehe unten). Die nächsten drei Symbole zeigen an, dass es sich auch um ein von Experten kuratiertes Gen handelt, dass Experimente zur Insertionsmutagenese bei Mäusen belegen, dass BRAF ein Krebsgen ist, und schließlich, dass BRAF ein „Cancer Hallmark“-Gen ist. Nach diesen Symbolen finden Sie viele weitere Details über BRAF, darunter Koordinaten, Synonyme, einen Link zu COSMIC-3D (siehe unten) und vieles mehr.
Wenden wir uns nun der Genansicht zu. Das Histogramm der Mutationshäufigkeit (Substitution) zeigt einen sehr dramatischen „Hotspot“ von Mutationen an Position 600 (z.B. p.V600E). Bewegen Sie die Maus über diesen Teil des Histogramms, um Details zu sehen. Dies ist eine sehr bekannte Treibermutation bei mehreren Krebsarten.
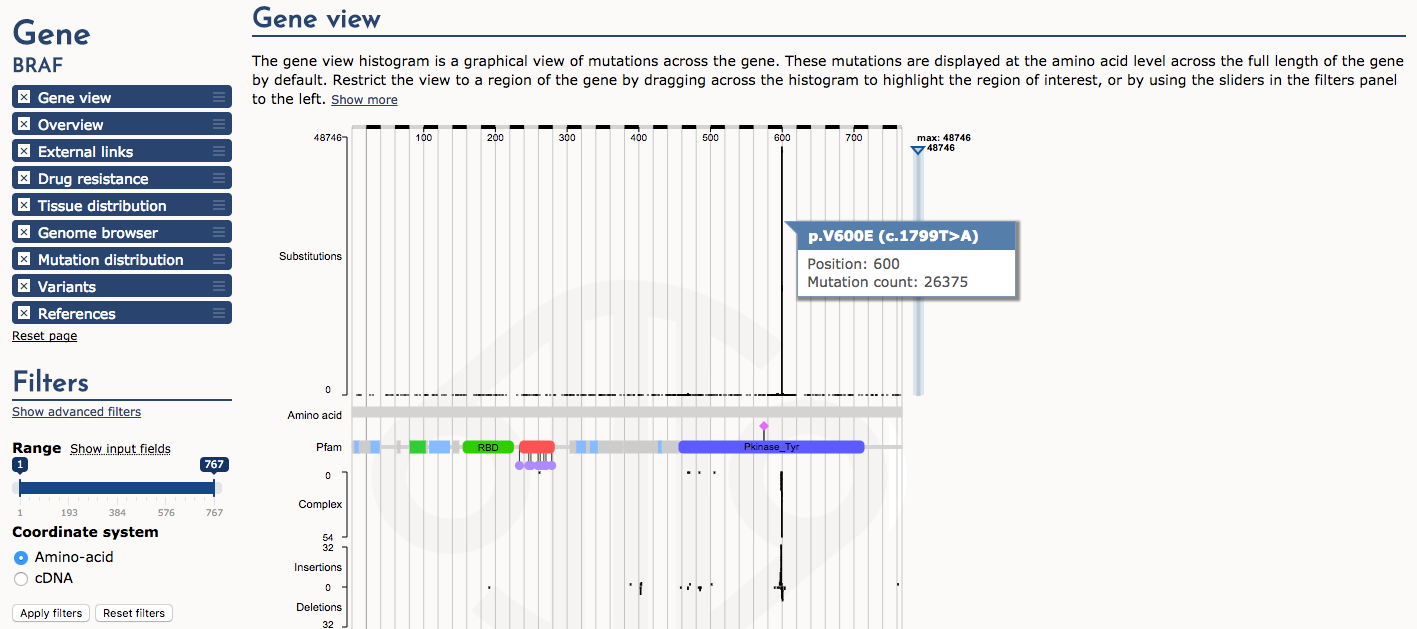
Welcher Teil (Domäne) des BRAF-Proteins ist von der p.V600E-Mutation betroffen?
Die Protein-Tyrosinkinase-Domäne (Pfam)
Navigieren Sie schließlich zum Abschnitt „Gewebeverteilung“. Sortieren Sie die Tabelle nach „Punktmutationen“ -> „% mutiert“. Es fällt auf, dass Schilddrüsenkrebs und Hautkrebs (z. B. Melanom) bei weitem am häufigsten am BRAF-Genlocus mutiert sind (Anmerkung: NS bedeutet nicht angegeben). Eine Teilmenge der Proben weist auch eine erhöhte Kopienzahlvariation (CNV) und eine hochregulierte Expression auf. Im Allgemeinen neigen bestimmte, überwiegend mutierte Gene dazu, mit Krebsarten bestimmter Herkunft in Verbindung gebracht zu werden. Es gibt jedoch viele Ausnahmen von dieser Aussage, und einige Gene (z. B., TP53) sind bei vielen verschiedenen Krebsarten mutiert.
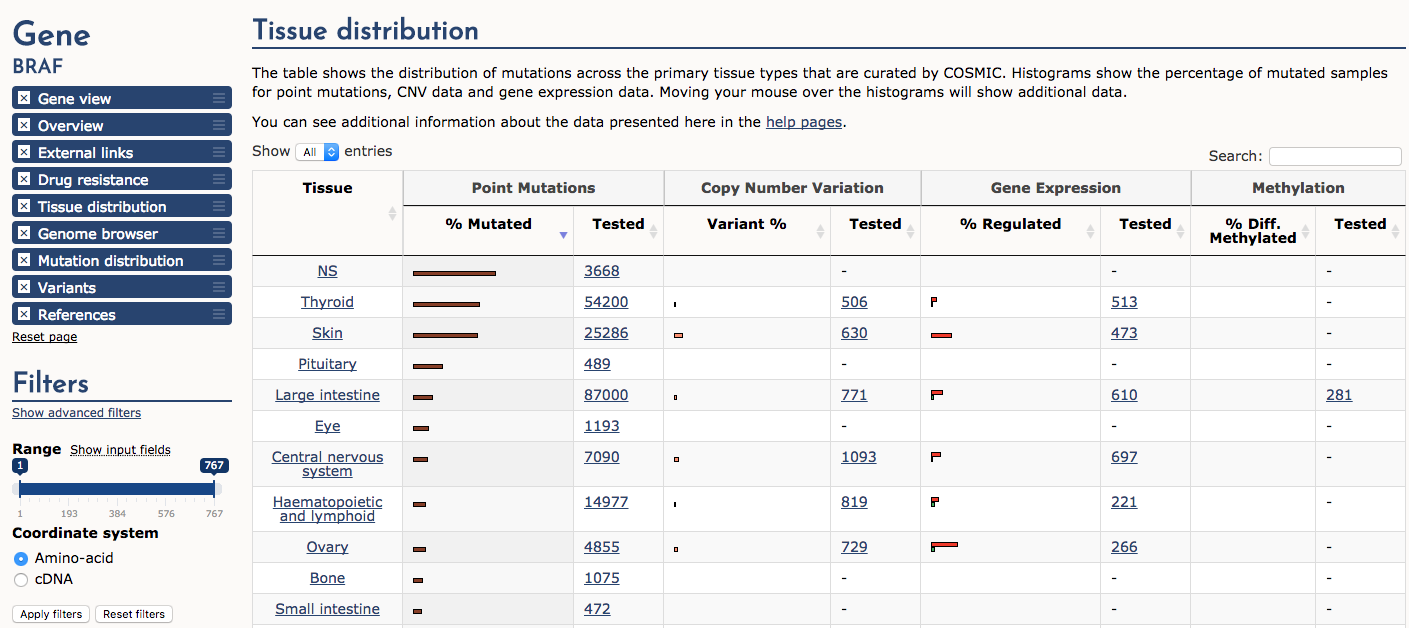
Welcher Krebsgewebetyp ist am häufigsten von einer BRAF-Überexpression betroffen?
Ungefähr 15 % der Eierstockkrebsfälle sind von einer BRAF-Überexpression betroffen
Der Krebsgen-Zensus
Der Krebsgen-Zensus (CGC) ist ein fortlaufender Versuch, die Gene zu katalogisieren, bei denen Mutationen (somatisch oder Keimbahn) kausal mit Krebs in Verbindung gebracht wurden. Die ursprüngliche Zählung und Analyse wurde von Futreal et al. 2004 in Nature Reviews Cancer veröffentlicht und wird weiterhin regelmäßig aktualisiert. Der CGC gilt weithin als endgültige Liste von Krebsgenen (Tumorsuppressoren und Onkogene). Navigieren Sie zum Cancer Gene Census über das Dropdown-Menü „Projekte“, das Sie auf jeder COSMIC-Seite finden. Diese Seite ist in drei Hauptabschnitte unterteilt: Krebsgenzählung, Aufschlüsselung und Abkürzungen. Im ersten Abschnitt wird eine einfache Tabelle mit allen Genen der Krebsgenzählung angezeigt.
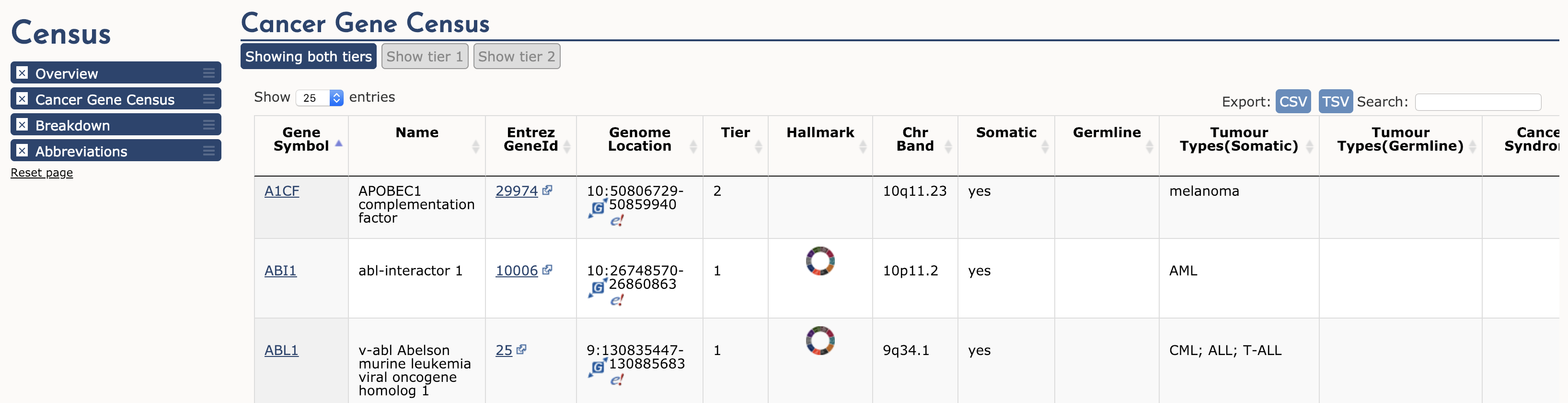
Zur Veranschaulichung betrachten wir ABL1 (das dritte Gen in der Tabelle). Die ersten vier Spalten enthalten den beschreibenden Namen des Gens, Links zu seinen COSMIC- und Entrez-Genseiten sowie die genomische Region mit Links zu COSMIC- und Ensembl-Browser-Ansichten. In den anderen Spalten erfahren wir, dass ABL1 auf dem Chromosomenband 9q34.1 liegt, von dem bekannt ist, dass es bei CML, ALL und T-ALL somatisch mutiert ist. Es wirkt als Onkogen und als Genfusionspartner. Tatsächlich ist ABL1 ein Teil der vielleicht berühmtesten Krebsfusion, BCR-ABL, dem Produkt des Philadelphia-Chromosom-Rearrangements. Diese Fusion definiert und steuert fast alle Fälle von CML, und ihre Entdeckung führte zu einem der erfolgreichsten Beispiele für eine gezielte Therapie (Imatinib). Um mehr über die Rolle von ABL1 bei Krebs zu erfahren, wählen Sie das Symbol ‚Census Hallmark‘. Ein „Hallmark“ ist ein Verweis auf die bahnbrechende Arbeit von Hanahan und Weinberg (2000), die 2011 aktualisiert wurde und in der sie vorschlugen, dass alle Krebsarten sechs (jetzt zehn) gemeinsame Merkmale (Hallmarks) aufweisen, die die Umwandlung von normalen Zellen in bösartige Krebszellen erklären. Die Kuratoren des Cancer Gene Census haben versucht, jedes Krebsgen im Census einem oder mehreren dieser Merkmale zuzuordnen. Die Grafik zeigt, welche Hallmarks durch ABL1 gefördert (grüne Balken) oder unterdrückt (blaue Balken) werden. In diesem Fall wird angenommen, dass ABL1 die proliferative Signalübertragung, die Veränderung der zellulären Energetik, die Instabilität des Genoms, die Angiogenese, die Invasion und die Metastasierung fördert und den programmierten Zelltod unterdrückt.
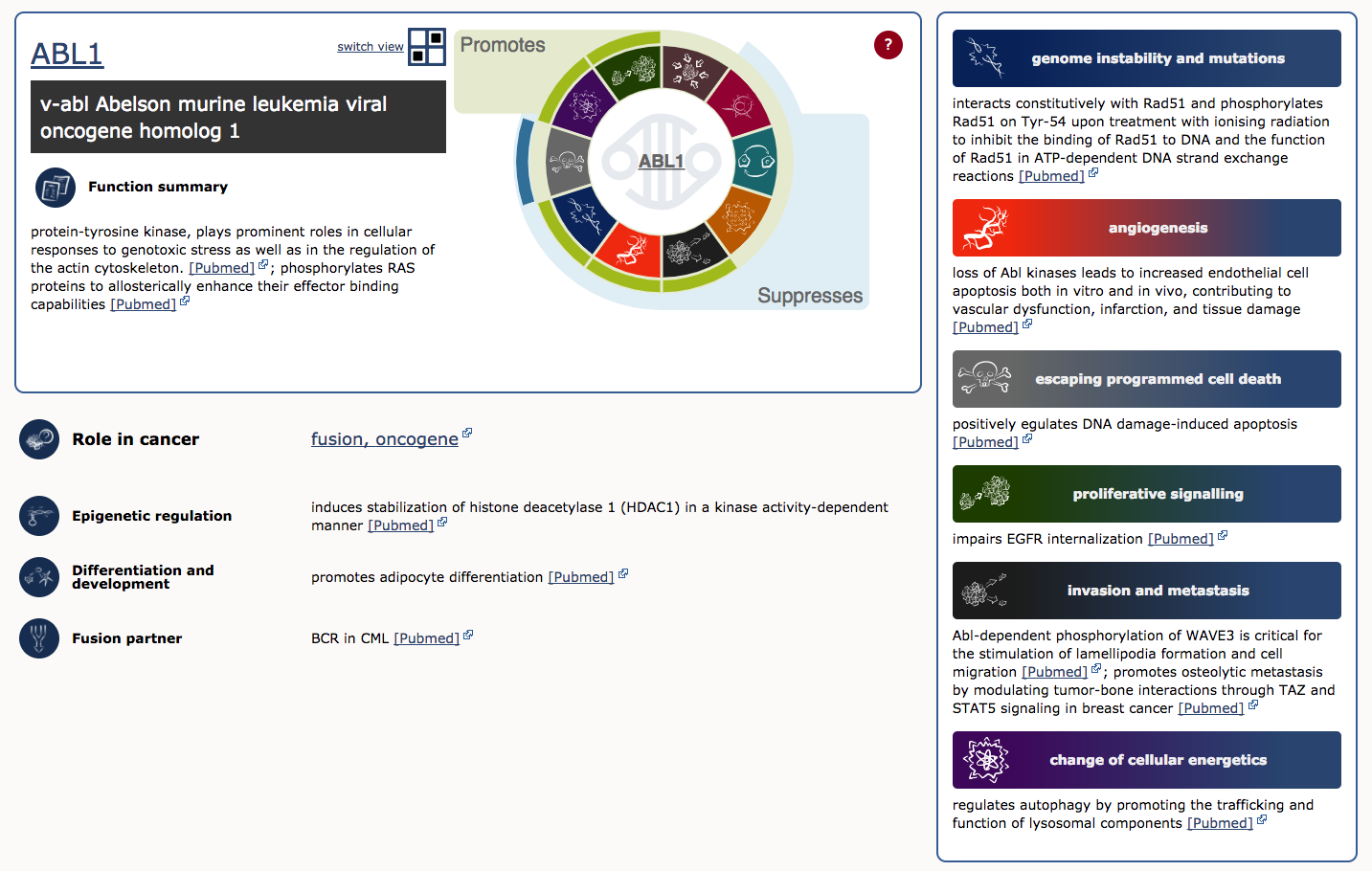
Wie viele Tumorsupressorgene (TSGs) und Onkogene gibt es im CGC?
Zum Zeitpunkt der Erstellung dieses Berichts gab es 315 TSGs und 315 Onkogene im CGC. Interessanterweise gibt es 72 Gene, die sowohl als Onkogen als auch als TSG fungieren.
Hinweis: Sie müssen sich bei COSMIC registrieren, um den vollständigen Cancer Gene Census herunterladen zu können. Sie könnten dann diese Datei in R laden und die Linux-Kommandozeile oder einen anderen Ansatz verwenden, um die Liste der TSGs und Onkogene zu ermitteln, die derzeit im CGC dokumentiert sind.