Einführung
Die reibungslose und effiziente Synthese biologisch aktiver Moleküle ist eines der wichtigsten Ziele der organischen und medizinischen Chemie. In den letzten Jahren haben sich Mehrkomponentenreaktionen zu einem wichtigen Werkzeug für die Synthese strukturell vielfältiger chemischer Bibliotheken von arzneimittelähnlichen polyfunktionellen organischen Molekülen entwickelt. Darüber hinaus bieten MCRs in mehrfacher Hinsicht den Vorteil der Einfachheit und synthetischen Effizienz gegenüber herkömmlichen chemischen Reaktionen. MCRs ermöglichen den Aufbau kombinatorischer Bibliotheken komplexer organischer Moleküle für eine effiziente Identifizierung und Optimierung von Leitstrukturen bei der Entdeckung von Arzneimitteln.
In Fortsetzung unserer laufenden Forschungsarbeiten zur mikrowellenunterstützten Synthese von Nanomaterialien haben wir festgestellt, dass nanokristalline Metalloxide aufgrund ihrer hohen katalytischen Aktivität und ihrer Wiederverwendbarkeit bei synthetischen und medizinischen Chemikern große Aufmerksamkeit erregt haben. Zinkoxid ist ein preiswertes, feuchtigkeitsstabiles, wiederverwendbares, handelsübliches und ungiftiges Material, das sowohl in polaren als auch in unpolaren Lösungsmitteln unlöslich ist. Es eignet sich für eine Vielzahl organischer Reaktionen wie Beckmann-Umlagerungen, N-Benzylierung, Acylierung, Dehydratisierung von Oximen, nukleophile Ringöffnungsreaktionen von Epoxiden, Synthese von cyclischem Harnstoff und N-Formylierung von Aminen. Insbesondere kristalline nano-ZnO-Oxide zeigen eine bessere katalytische Aktivität als ihre Gegenstücke in Bulk-Größe.
In den letzten Jahren wurde der Synthese von Dihydropyridinverbindungen aufgrund ihrer enormen Anwendung in verschiedenen Forschungsbereichen, einschließlich der biologischen Wissenschaft und der medizinischen Chemie, viel Aufmerksamkeit gewidmet. Viele DHPs sind bereits im Handel erhältlich, wie z. B. Amlodipin, Felodipin, Isradipin, Lacidipin, Nicardipin, Nitrendipin, Nifedipin und Nimodipin B, von denen Nitrendipin und Nemadipin B eine starke Kalziumkanalblockade aufweisen (Abbildung 1) und zu einer der wichtigsten Arzneimittelklassen für die Behandlung von Herz-Kreislauf-Erkrankungen geworden sind. Darüber hinaus besitzen Dihydropyridin-Derivate eine Vielzahl von biologischen Aktivitäten wie geroprotektive, hepatoprotektive, anti-atherosklerotische, antitumorale und antidiabetische Aktivitäten. Weit verbreitete Studien haben ergeben, dass Dihydropyridineinheiten enthaltende Verbindungen verschiedene medizinische Funktionen aufweisen, wie z. B. neuroprotektive, plättchenaggregationshemmende Aktivität, zerebrale Anti-Ischämie-Aktivität bei der Behandlung der Alzheimer-Krankheit, Chemosensibilisatoren in der Tumortherapie. Medikamentenresistenz-Modifikatoren, Antioxidantien und ein Medikament zur Behandlung von Harninkontinenz.
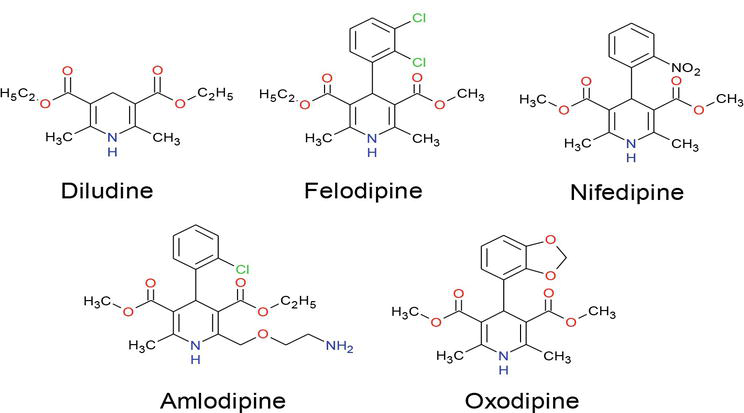
Abbildung 1.
Medikamente, die 1,4-DHP-Anteile enthalten.
Um diese biologischen Eigenschaften zu modellieren und zu verstehen und um neue chemotherapeutische Wirkstoffe auf der Grundlage von 1,4-DHP-Verbindungen zu entwickeln, wurden erhebliche Anstrengungen unternommen, um wirksame Methoden für ihre Synthese zu entwickeln. Im Allgemeinen wurden 1,4-DHP durch die Hantzsch-Methode synthetisiert, die eine Zyklokondensation eines Aldehyds, eines β-Ketoesters und Ammoniak entweder in Essigsäure oder unter Rückfluss in Alkoholen für lange Reaktionszeiten beinhaltet, was typischerweise zu geringen Ausbeuten führt. Andere Methoden umfassen die Verwendung von Mikrowellen, hohen Temperaturen unter Rückfluss, Organokatalysatoren und Metalltriflaten.
In jüngster Zeit ist die DNA ein wichtiger Angriffspunkt für Medikamente und sie reguliert viele biochemische Prozesse, die im zellulären System stattfinden. Die Wechselwirkungen kleiner Moleküle mit der DNA werden nach wie vor intensiv und umfassend auf ihre Nützlichkeit als Sonden für die zelluläre Replikation und Transkriptionsregulierung sowie auf ihr Potenzial als Arzneimittel untersucht. Insbesondere die Entwicklung von Verbindungen auf der Grundlage ihrer Fähigkeit, DNA zu spalten, ist nicht nur aus primär biologischer Sicht von großer Bedeutung, sondern auch im Hinblick auf einen photodynamischen therapeutischen Ansatz zur Entwicklung wirksamer Arzneimittel. 1,4-Dihydropyridin-Derivate haben aufgrund ihrer vielfältigen biologischen Anwendungen die Aufmerksamkeit der Chemiker auf sich gezogen. Die biologische Bedeutung dieser Verbindungsklasse hat uns dazu veranlasst, diese Reihe zu erweitern, indem wir uns mit der Synthese und der DNA-Photospaltung von 1,4-Dihydropyridin-Derivaten beschäftigt haben. In dieser Mitteilung wird über die Synthese von 1,4-Dihydropyridin-Derivaten und ihre DNA-Photospaltung sowie über molekulares Docking berichtet.
In der Literatur sind mehrere Methoden zur Synthese von 1,4-Dihydropyridin-Derivaten bekannt. In Fortsetzung unseres Programms zur Chemie von Nanomaterialien berichten wir hier über eine effiziente Mikrowellenmethode für die Synthese von kristallinen ZnO-NPs. Das in dieser Arbeit verwendete ZnO wurde nach einer modifizierten Methode synthetisiert. Die hergestellten kristallinen ZnO-Nanopartikel wurden mittels XRD, SEM und EDX charakterisiert (Abbildung 2). Unser synthetischer Ansatz begann mit der Kondensation von 1 Äquivalent Benzaldehyd 1a mit 2 Äquivalenten Ethylacetoacetat 2a und 2 Äquivalenten NH4OAc 3a in Gegenwart von ZnO-Nps und führte zur Bildung von Hantzsch 1,4-Dihydropyridin 4a (Abbildung 3). Die Reaktion war unter Mikrowellenbestrahlung in 5 min abgeschlossen, und das Produkt wurde durch die übliche Aufarbeitung in 90 % Ausbeute und hoher Reinheit isoliert. Unter ähnlichen Bedingungen reagierten verschiedene substituierte aromatische Aldehyde, die entweder elektronenabgebende oder -abziehende Substituenten tragen, mit 1,3-Diketonen unter Bildung von 1,4-DHPs in guten bis ausgezeichneten Ausbeuten, und die Ergebnisse sind in Tabelle 1 zusammengefasst.
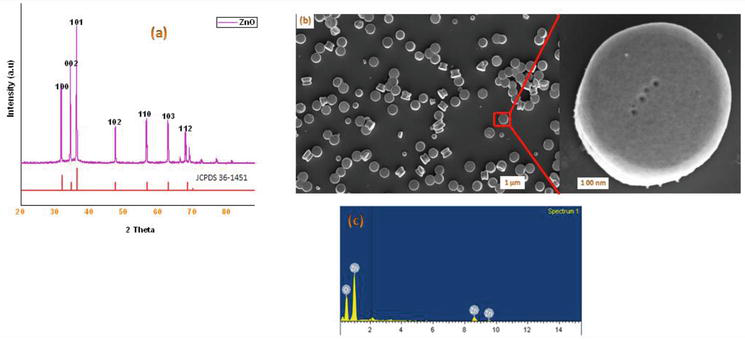
Abbildung 2.
(a) Pulver-XRD von erhaltenen ZnO-Nanopartikeln durch Mikrowellenmethode; (b) SEM-Bilder von ZnO-NPs; (c) EDX-Analysespektrum von erhaltenen ZnO-Nanopartikeln durch Mikrowellenmethode.
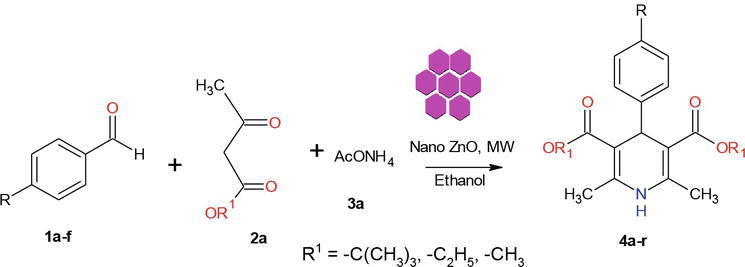
Abbildung 3.
Synthese von 1,4-Dihydropyridinen.
| Entrya | R | R1 | Produkte | Eintraga | Ausbeute (%)b |
|---|---|---|---|---|---|
| 1 | C6H5 | t-Bu | 4a | 1 | 90 |
| 2 | 4-MeO-C6H5 | t-Bu | 4b | 2 | 95 |
| 3 | 4-OH-C6H5 | t-Bu | 4c | 3 | 95 |
| 4 | 4-F-C6H5 | t-Bu | 4d | 4 | 95 |
| 5 | 4-Cl-C6H5 | t-Bu | 4e | 5 | 90 |
| 6 | 4-NO2-.C6H5 | t-Bu | 4f | 6 | 95 |
| 7 | C6H5 | Et | 4 g | 7 | 90 |
| 8 | 4-MeO-C6H5 | Et | 4 h | 8 | 95 |
| 9 | 4-OH-C6H5 | Et | 4i | 9 | 92 |
| 10 | 4-F-C6H5 | Et | 4j | 10 | 92 |
| 11 | 4-Cl-C6H5 | Et | 4 k | 11 | 90 |
| 12 | 4-NO2-C6H5 | Et | 4 l | 12 | 90 |
| 13 | C6H5 | Me | 4 m | 13 | 90 |
| 14 | 4-MeO-C6H5 | Me | 4n | 14 | 87 |
| 15 | 4-OH-C6H5 | Me | 4o | 15 | 90 |
| 16 | 4-F-C6H5 | Me | 4p | 16 | 90 |
| 17 | 4-Cl-C6H5 | Me | 4q | 17 | 90 |
| 18 | 4-NO2-C6H5 | Me | 4r | 18 | 90 |
Tabelle 1.
Synthese von 1,4-Dihydropyridinen.
aAlle Produkte wurden durch 1H-NMR- und 13C-NMR-Untersuchungen charakterisiert und mit den Literaturwerten verglichen. bAusbeuten der isolierten Produkte
Ein mikrowellenbestrahlungsunterstützter Prozess minimiert sehr oft die Bildung von Nebenprodukten und benötigt viel weniger Zeit als thermische Methoden. Die Hauptvorteile der Durchführung von Reaktionen unter kontrollierten Bedingungen in geschlossenen Gefäßen sind die signifikanten Geschwindigkeitssteigerungen und die höheren Produktausbeuten, die häufig erzielt werden können. In Fortsetzung unserer Studien zur Mikrowellensynthese von Nanomaterialien haben wir daher versucht, ein schnelles, mikrowellengestütztes Protokoll für die Synthese von 1,4-DHP unter Verwendung eines kristallinen ZnO-Nanokatalysators zu entwickeln (Abbildung 3).
Die DNA-Spaltung von 1,4-DHP-Derivaten wurde durch Agarosegel-Elektrophorese untersucht. Wenn zirkuläre Plasmid-DNA der Elektrophorese unterzogen wurde, wurde eine relativ schnelle Migration für die intakte, supergewickelte DNA (Typ I) beobachtet. Wenn ein Strang gespalten wird (Nicking), entspannt sich die supergewickelte DNA und bildet eine sich langsamer bewegende offene zirkuläre Form (Typ II). Wenn beide Stränge gespalten werden, entsteht eine lineare Form (Typ III), die zwischen Typ I und Typ II wandert. Die Umwandlung von Typ I (supercoiled) in Typ II (nicked circular) wurde mit unterschiedlichen Konzentrationen von 1,4-DHP beobachtet und 2 Stunden lang in 1:9 DMSO/Trisbuffer (20 μM, pH 7,2) bei 365 nm bestrahlt. Bei der Kontrolle, in der kein 1,4-DHP vorhanden war (Spur 1), wurde keine DNA-Spaltung beobachtet (Abbildung 4). Mit zunehmender Konzentration dieser 1,4-DHP nahm die Menge des Typs I der pUC 19-DNA allmählich ab, während der Typ II zunahm (Abbildung 4).
Bei einer Konzentration von 40 μM kann die Verbindung (4c) nur eine 30 %ige Umwandlung der DNA vom Typ I in den Typ II fördern (Abbildung 5). Bei einer Konzentration von 80 μM kann die Verbindung (4c) fast 80 % der DNA vom Typ I in II umwandeln (Abbildung 5). Das Spaltungspotenzial der Testverbindungen wurde durch den Vergleich der Banden, die in der Kontrolle und den Testverbindungen bei einer Konzentration von 80 μM auftraten, bewertet. Andere Derivate zeigten jedoch eine viel geringere Spaltungseffizienz für pUC 19-DNA. Selbst bei einer Konzentration von 80 μM können nur 40 % der DNA vom Typ I in den Typ II umgewandelt werden (Abbildung 5).
Bei höheren Konzentrationen um 130 μM werden die Verbindungen jedoch ausgefällt und es gibt kein Moment in der DNA. Das Bild (Abbildung 6) zeigt deutlich, dass die Verbindungen (4b, 4c, 4d, 4e, 4f und 4 g) bei einer Konzentration von 80 μM eine DNA-Spaltung der pUC19-DNA zeigen. Die Ergebnisse deuten darauf hin, dass Verbindungen, die -OCH3 und -OH an der -para-Position des Phenylrings (C-6) tragen, die DNA vollständig spalten, während andere Verbindungen eine fast vollständige Spaltung der DNA zeigen. Insgesamt deutet dies darauf hin, dass die Alkoxygruppen hochreaktive Radikale sind, die effizient Wasserstoffatome an C-4′ der 2-Desoxyribose abspalten. Es ist interessant festzustellen, dass die Hydroxylgruppe Berichten zufolge in Gegenwart von Fotostrahlung eine durch Sauerstoffradikale vermittelte DNA-Schädigung hervorruft.
Die Studien zur Struktur-Aktivitäts-Beziehung von 1,4-DHP im Hinblick auf Studien zur DNA-Fotospaltung zeigen, dass die Änderungen des Substitutionsmusters an den Positionen C-3, C-4 und C-5 den 1,4-DHP-Ring verändern. Der Osiris Property Explorer ist ein solches wissensbasiertes Tool zur Vorhersage der Aktivität, das die Wahrscheinlichkeit von Arzneimitteln, die Bewertung von Arzneimitteln und unerwünschte Eigenschaften wie mutagene, tumorigene, reizende und reproduktive Wirkungen neuartiger Verbindungen auf der Grundlage von chemischen Fragmentdaten verfügbarer Arzneimittel und Nichtarzneimittel vorhersagt (Tabelle 2). Es wurde festgestellt, dass die Verbindungen mit aliphatischen Gruppen wie -CH3, -COOCH3, -COOC2H5 und -COOC(CH3)3, die an C-2 und C-3 von 1,4-DHP gebunden sind, eine gute Aktivität aufwiesen. Andere Derivate, die einen elektronenabgebenden Substituenten wie eine Hydroxy- oder Methoxygruppe am Phenylring (C-6) besitzen, erhöhen die DNA-Fotospaltungsaktivität. Ein einsames Elektronenpaar am Sauerstoffatom der Methoxygruppe delokalisiert in den π-Raum des Benzolrings und erhöht dadurch die Aktivität. In ähnlicher Weise verringern elektronenziehende Substituenten wie 4-Fluorphenyl und 4-Chlorphenyl von 1,4-DHP die Aktivität. Diese Ergebnisse weisen darauf hin, dass die Alkoxysubstituenten und der Stickstoff des Pyridinrings in der 1,4-DHP-Struktur für die DNA-Spaltung verantwortlich sind.
Um die beobachteten spektroskopischen Ergebnisse zu rationalisieren und einen besseren Einblick in die Interkalationsmodalität zu erhalten, wurden die 1,4-DHP (4a-r) nacheinander an den DNA-Duplex der Sequenz d(CGCGAATTCGCG)2 dodecamer (PDB ID: 1BNA) angedockt, um die gewählte Bindungsstelle zusammen mit der bevorzugten Ausrichtung des Liganden innerhalb der DNA-Nebenrille vorherzusagen. Alle synthetisierten 1,4-DHP-Derivate wurden in ChemSketch gezeichnet und die Strukturen wurden im .mol-Format gespeichert. Anschließend wurde das .mol-Format in Hyperchem-7 verwendet, um die Fragmente anzupassen, gefolgt von einer Minimierung der Gesamtenergie der Liganden, damit sie eine stabile Konformation erreichen können, und die Datei wurde im .pdb-Format gespeichert.
Die 3D-Proteinstruktur der B-DNA wurde aus der RCSB PDB (einem Informationsportal für biologische makromolekulare Strukturen) entnommen. Die Wassermoleküle wurden aus der Datei entfernt, und das Protein wurde in 3D protoniert, um polare Wasserstoffatome hinzuzufügen. Die Bindungstasche wurde mit dem Site Finder identifiziert, und die entsprechenden Reste wurden ausgewählt. Die Docking-Parameter wurden auf die Standardwerte und den Scoring-Algorithmus eingestellt, die Docking-Läufe wurden auf 30 Konformationen pro Ligand beschränkt. Die angedockten Proteinstrukturen wurden im .pdb-Format gespeichert, und die Konformationen der Liganden wurden nacheinander untersucht. Die Komplexe mit den besten Konformationen wurden auf der Grundlage der höchsten Punktzahl, der niedrigsten Bindungsenergie und der minimalen RMSD-Werte ausgewählt.
Die synthetisierten organischen Verbindungen entfalten ihre biologische Aktivität effizienter, indem sie das jeweilige Protein oder die DNA an ihrer spezifischen Bindungsstelle binden. Die Identifizierung der mit den Liganden interagierenden Reste ist ein notwendiger Schritt auf dem Weg zu einem rationalen Arzneimitteldesign, zum Verständnis des molekularen Weges und der mechanistischen Wirkung des Proteins.
Molekulares Docking wurde zwischen dem starren Rezeptorprotein und den flexiblen Liganden durchgeführt. Tabelle 3 zeigt die Details der Docking-Ergebnisse einschließlich der RMSD- und Bindungsenergiewerte der Protein-Liganden-Komplexe. Die Liganden (4b, 4c, 4 h, 4i, 4n und 4o) binden stark an die B-DNA, wie aus ihren minimalen Bindungsenergiewerten hervorgeht, d. h. -13,8, -12,9 bzw. – 12,3 kcal/mol (Abbildung 7).
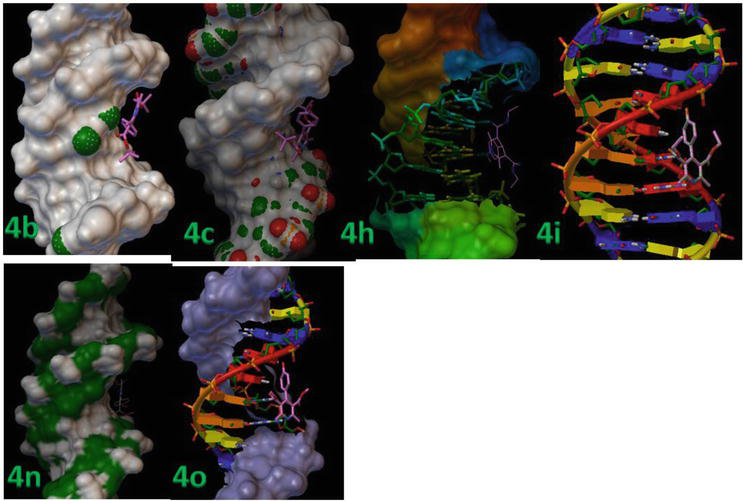
Abbildung 7.
1,4-DHP wurde nacheinander an den DNA-Duplex der Sequenz d(CGCGAATTCGCG)2 dodecamer (PDB ID: 1BNA) angedockt.
Abbildung 8 zeigt die Position der aktiven Stelle in der helikalen Struktur der DNA und es zeigt auch, dass alle angedockten Liganden innerhalb der Tasche gruppiert sind. Abbildung 8 zeigt die Wasserstoffbrückenbindungsinteraktion von 4c und 4d mit Schlüsselresten im aktiven Zentrum innerhalb der helikalen Struktur der DNA. In diesem Modell ist deutlich zu erkennen, dass die Verbindung 4c eine Wasserstoffbrückenbindung zwischen dem -OH und dem N1 von Thymin, d.h. DT7 und DT19, mit einer Bindungslänge von 2,02 bzw. 2,05 Ǻ bildet. Darüber hinaus bildeten die anderen 1,4-DHP-Derivate aufgrund der Ausrichtung des aromatischen Rings, der an den van-der-Waals-Wechselwirkungen beteiligt ist (Wireframe-Modell), und der flachen hydrophoben Bereiche der DNA-Bindungsstellen weniger H-Bindungswechselwirkungen mit der DNA (Tabelle 3). Diese Ergebnisse zeigten, dass die in silico molekularen Docking-Studien von 1,4-DHPs mit B-DNA darauf hindeuten, dass 1,4-DHPs das Potenzial besitzen, hydrophobe und H-Bindungs-Wechselwirkungen zu stören und dadurch die Stabilität der Bindung von B-DNA zu beeinträchtigen, und dass sie für Krebszelllinien wirksam sein können.
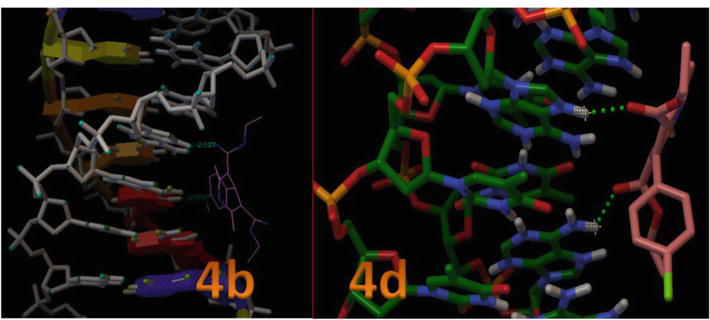
Abbildung 8.
Interaktion von 1,4-DHP mit DNA-Duplex der Sequenz d(CGCGAATTCGCG)2 dodecamer (PDB ID: 1BNA).