COSMIC, il Catalogue Of Somatic Mutations In Cancer, ospitato dal Wellcome Trust Sanger Institute, è una delle risorse più grandi e complete per esplorare l’impatto delle mutazioni somatiche nel cancro umano. È il prodotto di un massiccio sforzo di cura dei dati da parte di esperti, che presenta dati provenienti da migliaia di pubblicazioni e molti importanti set di dati di genomica del cancro su larga scala. COSMIC funge da portale principale per almeno quattro importanti progetti o risorse: COSMIC – il catalogo delle mutazioni somatiche nel cancro, il Cancer Gene Census, il Cell Lines Project e COSMIC-3D. Gli utenti accademici possono utilizzare e scaricare (con registrazione) i dati gratuitamente, a condizione di non ridistribuire i dati e di accettare i termini della licenza. Gli utenti a scopo di lucro devono pagare una tassa per scaricare COSMIC. Queste risorse forniscono l’accesso a una serie molto ricca di dati, visualizzazioni e interpretazioni per comprendere le mutazioni del cancro e i geni del cancro.
Il database COSMIC
Per esplorare COSMIC si può semplicemente navigare alla pagina principale e cercare un gene, un tipo di cancro, una mutazione, ecc nella casella di ricerca. Per illustrare esploreremo i risultati per un singolo gene. Digita BRAF nell’interfaccia di ricerca e premi invio. Al momento della scrittura, questo termine di ricerca ha restituito risultati per 1 gene, 826 mutazioni, 49 tumori, 226 campioni, 1575 citazioni Pubmed e 0 studi (vedi schede dei risultati). Si noti che COSMIC contiene risultati per quasi 300.000 campioni testati e oltre 50.000 mutazioni in BRAF.
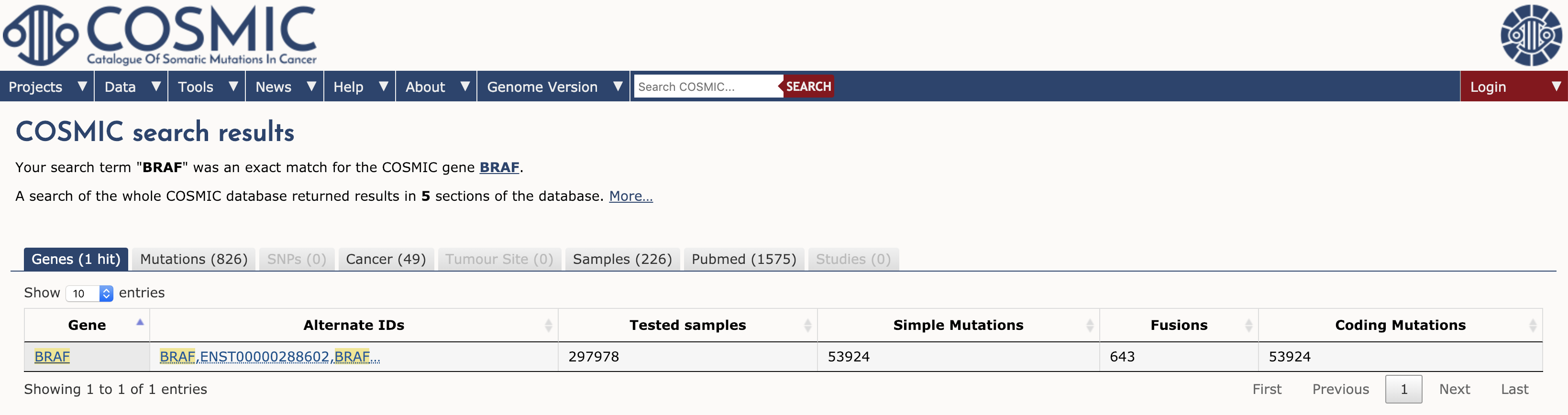
Se selezioniamo il risultato BRAF, COSMIC restituisce una pagina dettagliata che fornisce: riassunti dei geni, collegamenti ad altre risorse COSMIC (ad esempio, geni Census, geni Hallmark, ecc), collegamenti esterni, resistenza ai farmaci, distribuzione dei tessuti, vista browser genoma, distribuzione delle mutazioni, varianti e riferimenti. Guarderemo alcune di queste sezioni. Per prima cosa, guardiamo la sezione Panoramica. Lungo la parte superiore di questa sezione ci sono diverse icone utili. L’icona ‘gene del censimento’ ci dice che BRAF è un gene del cancro noto secondo il censimento dei geni (vedi sotto). Le prossime tre icone ci dicono che è anche un ‘Expert curated gene’, che gli esperimenti di mutagenesi inserzionale nel topo supportano che BRAF è un gene del cancro, e infine che BRAF è un gene ‘Cancer Hallmark’. Dopo queste icone ci sono molti altri dettagli su BRAF tra cui coordinate, sinonimi, link a COSMIC-3D (vedi sotto), e altro ancora.
In seguito, esaminiamo la vista Gene. L’istogramma della frequenza di mutazione (sostituzione) mostra un “hotspot” molto drammatico di mutazioni in posizione 600 (ad esempio, p.V600E). Passa il mouse su questa parte dell’istogramma per vedere i dettagli. Questa è una mutazione driver molto nota in diversi tipi di cancro.
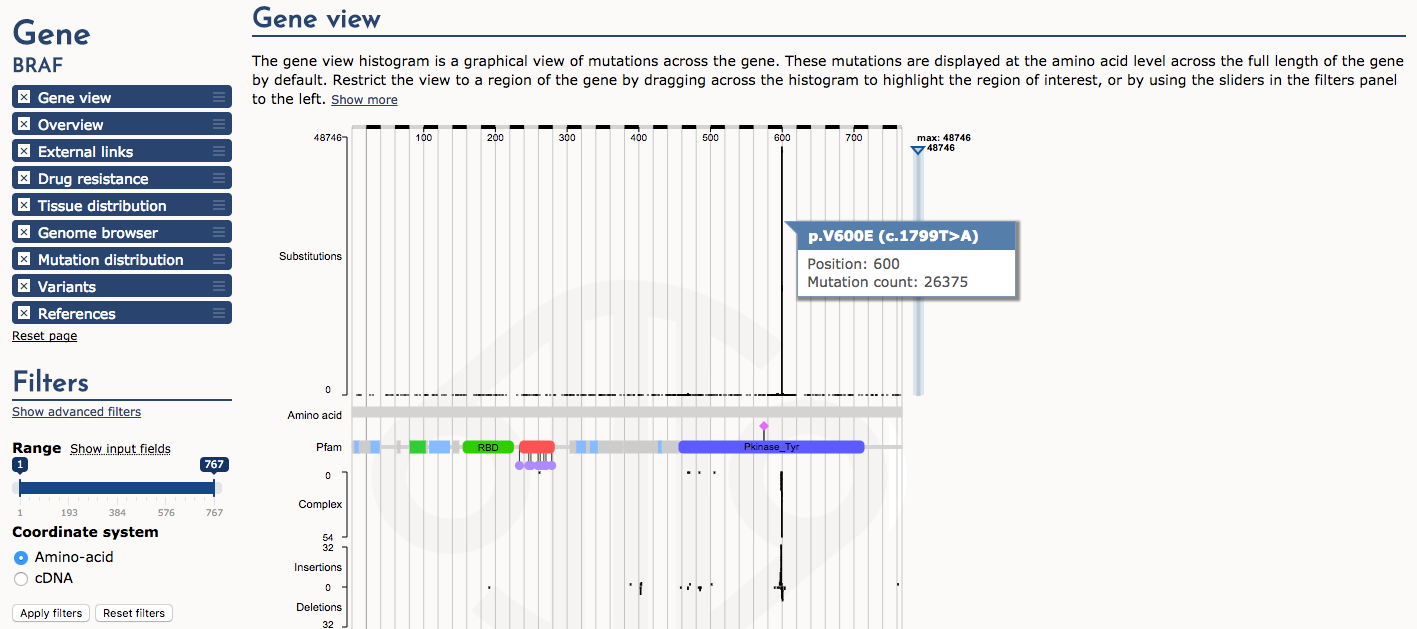
Quale parte (dominio) della proteina BRAF è interessata dalla mutazione p. V600E?V600E?
Il dominio della proteina tirosin-chinasi (Pfam)
Infine, passa alla sezione ‘Distribuzione dei tessuti’. Ordina la tabella per ‘Point mutations’ -> ‘% Mutated’. Si noti che i tumori della tiroide e della pelle (ad esempio, il melanoma) sono di gran lunga i più frequentemente mutati al locus del gene BRAF (nota NS significa non specificato). Un sottoinsieme di campioni mostra anche guadagni di variazione del numero di copie (CNV) ed espressione up-regolata. In generale, alcuni geni mutati in modo predominante tendono ad essere associati a tumori di determinate origini. Tuttavia, ci sono molte eccezioni a questa affermazione e alcuni geni (ad es, TP53) sono ampiamente mutati in molti tipi di cancro diversi.
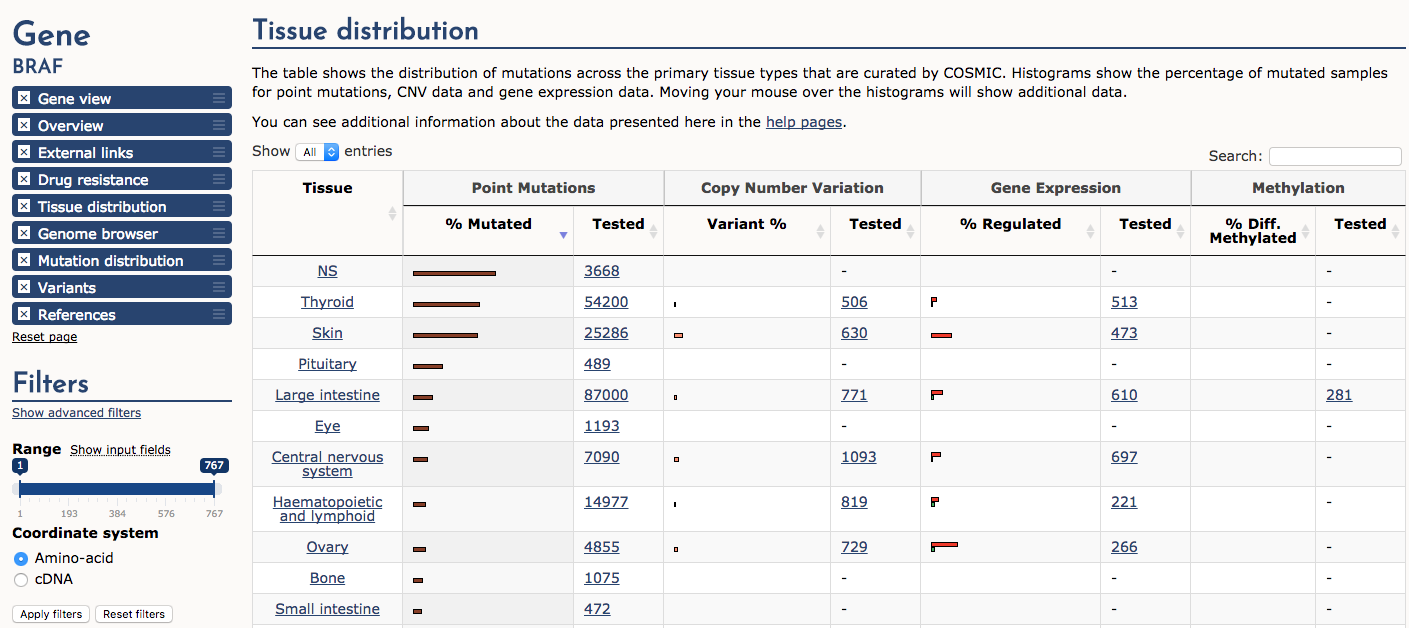
Quale tipo di tessuto tumorale è più comunemente colpito dalla sovraespressione di BRAF?
Circa il 15% dei tumori ovarici sono affetti da sovraespressione di BRAF
The Cancer Gene Census
Il Cancer Gene Census (CGC) è uno sforzo continuo per catalogare quei geni per i quali mutazioni (somatiche o germinali) sono state causalmente implicate nel cancro. Il censimento originale e l’analisi è stato pubblicato in Nature Reviews Cancer da Futreal et al. 2004 ma continua a ricevere aggiornamenti regolari. Il CGC è ampiamente considerato come un elenco definitivo dei geni del cancro (soppressori del tumore e oncogeni). Navigare al Cancer Gene Census dal menu a discesa ‘Projects’ disponibile su qualsiasi pagina COSMIC. Questa pagina è suddivisa in tre sezioni principali: Cancer Gene Census, Breakdown, e Abbreviazioni. Nella prima sezione, viene visualizzata una semplice tabella di tutti i geni Cancer Gene Census.
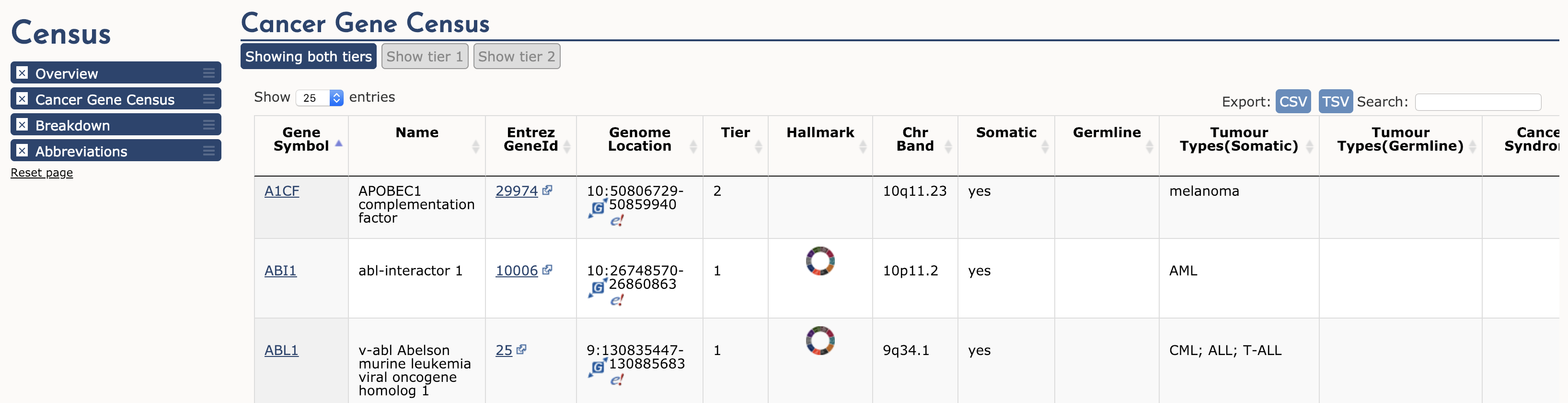
Per illustrare, esaminiamo ABL1 (il terzo gene nella tabella). Le prime quattro colonne forniscono il nome descrittivo del gene, i link alle sue pagine COSMIC e Entrez gene, e la regione genomica con i link alle viste COSMIC e Ensembl Browser. Altre colonne ci dicono che ABL1 si trova sulla banda cromosomica 9q34.1, nota per essere mutata somaticamente in CML, ALL e T-ALL. Agisce come oncogene e come partner di fusione genica. Infatti, ABL1 è una parte della fusione forse più famosa del cancro, BCR-ABL, il prodotto del riarrangiamento del cromosoma Philadelphia. Questa fusione definisce e guida quasi tutti i casi di CML, e la sua scoperta ha portato a uno dei più riusciti esempi di terapia mirata (Imatinib). Per saperne di più sul ruolo di ABL1 nel cancro seleziona l’icona ‘Census Hallmark’. Un ‘Hallmark’ è un riferimento al documento seminale di Hanahan e Weinberg (2000), aggiornato nel 2011, in cui hanno proposto che tutti i tumori condividono sei (ora dieci) tratti comuni (hallmark) che spiegano la trasformazione delle cellule normali in cellule tumorali maligne. I curatori del Cancer Gene Census hanno cercato di assegnare ogni gene del cancro nel censimento a uno o più di questi Hallmark. Il grafico mostra quali hallmark sono promossi (barre verdi) o soppressi (barre blu) da ABL1. In questo caso, si pensa che ABL1 promuova la segnalazione proliferativa, il cambiamento dell’energia cellulare, l’instabilità del genoma, l’angiogenesi, l’invasione e la metastasi, e sopprima la morte cellulare programmata.
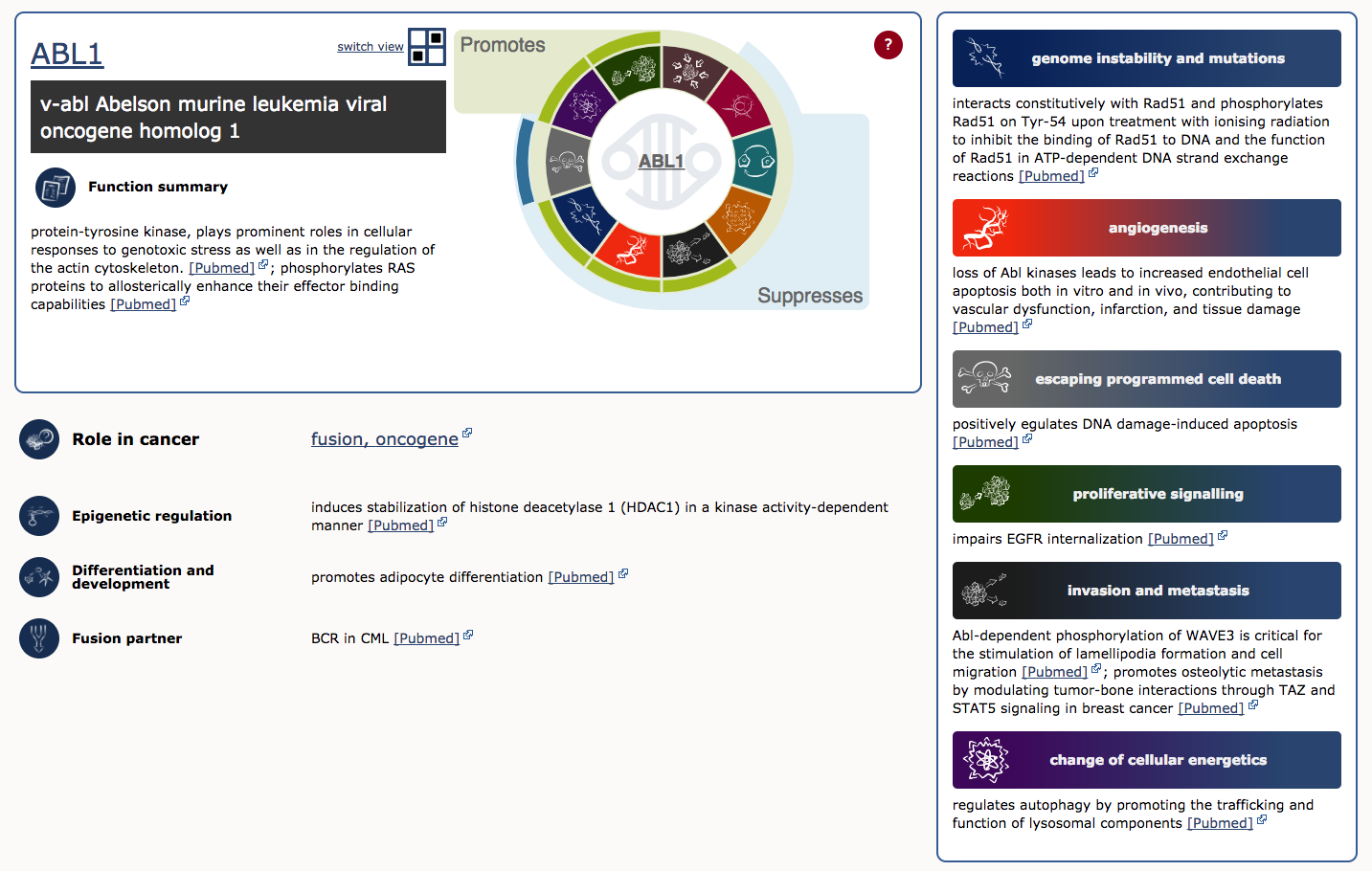
Quanti geni tumor supressor (TSGs) e oncogeni ci sono in CGC?
Al momento della scrittura c’erano 315 TSGs e 315 oncogeni in CGC. È interessante notare che ci sono 72 geni che funzionano sia come oncogene che come TSG.
Nota: È necessario registrarsi con COSMIC per scaricare il completo Cancer Gene Census. Si potrebbe quindi caricare questo file in R, e utilizzare la riga di comando linux, o qualche altro approccio per determinare l’elenco dei TSG e degli oncogeni attualmente documentati nel CGC.