Zwemmen in een zee van cellen: Waarom single-cell genomics?
Complexe biologische systemen zijn het resultaat van de gecoördineerde functie van individuele cellen die een ingewikkelde “dans” uitvoeren, waarbij elke cel zijn eigen speciale rol speelt in het geheel. Vanwege deze complexiteit, genexpressie onderzoek in organismen, weefsels of celpopulaties wordt vaak beperkt door het gebruik van traditionele bulk RNA-seq methoden. Als wetenschappers, we intuïtief begrijpen dat wanneer we “malen en vinden” om experimenten uit te voeren op onze verdunde mix van RNA, we echt alleen maar een klein idee van wat er gaande zou kunnen zijn in onze cellen van belang te krijgen: cel-tot-cel verschillen worden gemist en cellulaire heterogeniteit kan volledig worden gemaskeerd. In feite, bulk RNA expressie analyse beschrijft vaak een afgeleide toestand waarin geen (of zeer weinig) van de cellen daadwerkelijk bestaan!
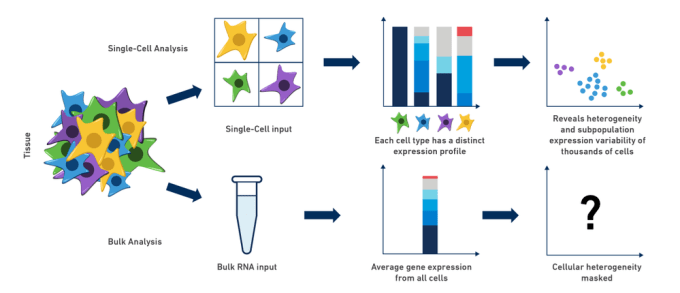
Misschien bent u een neurowetenschapper die in muizenhersenen werkt en de ontwikkeling bestudeert van een subpopulatie van neuronen die een specifieke neurotransmitter produceren. Als je die populatie maar zou kunnen isoleren en cel voor cel zou kunnen analyseren, zouden de potentiële inzichten inderdaad neuronaal stimulerend zijn! Hetzelfde geldt voor kankeronderzoek: hoe kun je de genexpressie en, wat misschien nog belangrijker is, de cellulaire heterogeniteit in een tumor het beste uit elkaar houden? Wat als je niet alleen inter-tumor, maar ook intra-tumor verschillen zou kunnen vergelijken en de immuuncelpopulaties in relatie tot de tumormicro-omgeving zou kunnen onderzoeken? Deze diversiteit, vaak verborgen door bulk RNA-seq methoden, kan worden onthuld door het gebruik van single cell RNA-seq om specifiek te verkennen deze klinisch belangrijke celpopulaties. De voorbeelden van nadelen van bulk analyse zijn er in overvloed, en ontleden celtype verschillen in de meeste, zo niet alle van deze complexe biologische systemen is van cruciaal belang voor ons begrip van hun bijdragen, vooral tijdens de ontwikkeling en in de progressie van disease.
Hoe isoleren en analyseren we enkele cellen?
10x Genomics ‘single-cell RNA-seq (scRNA-seq) technologie, de Chromium ™ Single Cell 3 ‘Solution, stelt u in staat om transcriptomen te analyseren op een cel-per-cel basis door het gebruik van microfluïdische partitionering om enkele cellen vast te leggen en te bereiden barcoded, next-generation sequencing (NGS) cDNA-bibliotheken. Specifiek, enkele cellen, omgekeerde transcriptie (RT) reagentia, Gel Beads met gebarcodeerde oligonucleotiden, en olie worden gecombineerd op een microfluïdische chip om reactie blaasjes genaamd Gel Beads in Emulsie, of GEMs te vormen. GEM’s worden parallel gevormd in de microfluïdische kanalen van de chip, waardoor de gebruiker 100’s tot 10.000’s afzonderlijke cellen kan verwerken in één enkele 7 minuten durende Chromium™ Instrument run. Het is belangrijk op te merken dat cellen worden geladen bij een beperkende verdunning om het aantal GEMs die een enkele cel bevatten te maximaliseren om een laag doublettarief te verzekeren, met behoud van een hoog celterugwinningspercentage van maximaal ~65%.
Elke functionele GEM bevat een enkele cel, een enkele Gel Bead, en RT-reagentia. Binnen elk GEM-reactieblaasje wordt een enkele cel gelyseerd, wordt de Gel Bead opgelost om de identiek gebarcodeerde RT-oligonucleotiden in oplossing vrij te maken, en vindt omgekeerde transcriptie van gepolyadenyleerd mRNA plaats. Dientengevolge zullen alle cDNA’s van een enkele cel dezelfde streepjescode hebben, waardoor de sequencing-lezingen kunnen worden teruggekoppeld naar hun oorspronkelijke enkele cellen van oorsprong. De voorbereiding van NGS-bibliotheken uit deze gebarcodeerde cDNA’s wordt vervolgens uitgevoerd in een zeer efficiënte bulkreactie. De video hieronder geeft een goede visuele uitleg van hoe dit allemaal werkt.
Van sample prep tot how-to video’s: Handige links voor aan de slag
De Chromium Single Cell 3′ workflow begint met uw cellen van belang, gevolgd door NGS-bibliotheekconstructie met behulp van onze reagens kits en de Chromium Controller. Bereiding van uw barcoded cDNA bibliotheek (zoals in het algemeen beschreven in de bovenstaande video) is een belangrijke stap en trouw aan het gezegde “garbage in, garbage out” – voorbereiding van hoge kwaliteit celsuspensies is de sleutel tot het krijgen van het meeste uit of uw RNA-seq gegevens. Als u hulp nodig met de cel bereidingsproces, hebben we meerdere gedemonstreerd protocollen beschikbaar op onze website, zoals dissociating neuraal weefsel of de voorbereiding mos protoplast suspensies voor gebruik in scRNA-seq, met meer protocollen worden regelmatig toegevoegd op onze support website. Voor begeleiding en aanbevelingen over hoe u het beste prep uw monsters zodra ze in suspensie, een goed uitgangspunt is onze single cell voorbereiding gids, waar u meer kunt leren over de beste praktijken en bekijk een algemeen protocol. Als u meer van een visuele leerling, onze how-to-video’s, met name de hoofdstukken 4 tot en met 7, moet u helpen lopen door het monster en de bibliotheek bereidingsproces.



-
Single Cell Prep Guide
-
Demonstrated Protocols
-
How-to-video’s
-
Chromium™ Controller
-
Gebruiksgidsen
-
Hoe-to-video’s
-
Cell Ranger™ Software
-
Loupe™ Cell Browser
-
How-to-video’s
Benieuwd naar uw gegevens, cel voor cel
Nadat uw bibliotheek is voorbereid, wordt de sequentiebepaling uitgevoerd met behulp van sequentiebepalingsinstrumenten van Illumina®. Dit leidt tot het leuke gedeelte: het verzamelen van biologische inzichten uit uw single cell RNA-seq data! De (potentieel) enorme hoeveelheid data die u genereert met de Chromium Single Cell 3′ Solution kan eenvoudig worden verwerkt door onze software en visualisatie tools die zijn ontworpen om veel van het giswerk weg te nemen-Cell Ranger™ Software transformeert de barcoded sequencing data in bestanden die klaar zijn voor single cell expressie analyse, en visualisatie wordt bereikt via de Loupe™ Cell Browser. Als u een stap-voor-stap tutorial wilt over het gebruik van onze software, behandelen de hoofdstukken 8 tot en met 11 van onze how-to-video’s alles van het tellen van transcripten met Cell Ranger Software tot prachtige visualisatie van gegevens met Loupe Cell Browser. Er zijn ook een groeiend aantal enkele cel analyse tools ontwikkeld door de gemeenschap, waaronder Seurat, Monocle en Cell View. Lees meer over deze analyse tools en andere single-cel artikelen op de 10x Genomics Blog, waar we belangrijke onderzoeksbijdragen benadrukken, tips en trucs geven, en u op de hoogte houden van alles wat single cell!
Als u andere vragen of zorgen heeft over het aan de slag gaan met de Chromium Single Cell 3 ‘Solution, voel je vrij om hier een reactie achter te laten of neem direct contact met ons op. Of het nu het bijhouden van de hersenen organoid ontwikkeling, het bestuderen van beenmergtransplantatie bij patiënten met acute myeloïde leukemie, of het ontrafelen van een nieuwe route voor intestinale stamcellen zelfvernieuwing, het aantal Chromium Single Cell publicaties groeien dagelijks, en we kijken ernaar uit om te horen over al uw verbazingwekkende onderzoek!