Úszás a sejtek tengerében:
A komplex biológiai rendszerek az egyes sejtek összehangolt működésének eredményeként jönnek létre, amelyek bonyolult “táncot” járnak, és mindegyikük sajátos szerepet játszik az együttesben. E komplexitás miatt a szervezetek, szövetek vagy sejtpopulációk génexpressziós kutatását gyakran korlátozza a hagyományos tömeges RNS-seq módszerek használata. Tudósként intuitív módon megértjük, hogy amikor “őrölünk és találunk”, hogy a hígított RNS-keverékünkön kísérleteket végezzünk, valójában csak egy kis képet kapunk arról, hogy mi történhet a minket érdeklő sejtekben: a sejtek közötti különbségek kimaradnak, és a sejtek heterogenitása teljesen elfedhető. Valójában az ömlesztett RNS-expressziós elemzések gyakran olyan feltételezett állapotot írnak le, amelyben valójában egyetlen sejt (vagy nagyon kevés) sem létezik!”
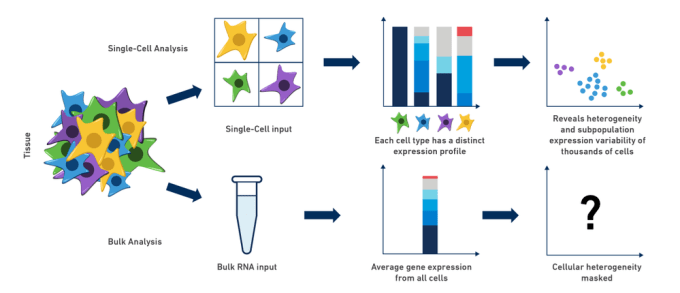
Talán Ön neurológus, aki egéragyban dolgozik, és egy bizonyos neurotranszmittert termelő neuronok egy alpopulációjának fejlődését tanulmányozza. Ha ezt a populációt el tudná különíteni és sejtről sejtre elemezni, a lehetséges felismerések valóban neuronálisan stimulálóak lennének! Hasonlóképpen a rákkutatás esetében: hogyan lehet a legjobban elkülöníteni a génexpressziót és, ami talán még fontosabb, a sejtek heterogenitását egy tumorban? Mi lenne, ha nemcsak a tumorok közötti, hanem a tumoron belüli különbségeket is össze lehetne hasonlítani, és fel lehetne tárni a tumor mikrokörnyezetéhez kapcsolódó immunsejt-populációkat? Ez a sokféleség, amelyet a tömeges RNS-seq módszerek gyakran elfednek, feltárható az egysejtes RNS-seq alkalmazásával, hogy kifejezetten ezeket a klinikailag jelentős sejtpopulációkat vizsgáljuk. A tömeges analízis hátrányaira rengeteg példa van, és a sejttípusbeli különbségek feltárása a legtöbb, ha nem az összes ilyen összetett biológiai rendszerben kritikus fontosságú a hozzájárulásuk megértéséhez, különösen a fejlődés során és a betegség progressziójában.
Hogyan izoláljuk és elemezzük az egyes sejteket?
A 10x Genomics egysejtes RNS-seq (scRNS-seq) technológiája, a Chromium™ Single Cell 3′ Solution lehetővé teszi a transzkriptomok sejtenkénti elemzését mikrofluidikus partícionálás alkalmazásával az egyes sejtek befogásához és vonalkódolt, következő generációs szekvenálási (NGS) cDNS-könyvtárak készítéséhez. Pontosabban, az egyes sejtek, a reverz transzkripciós (RT) reagensek, a vonalkódolt oligonukleotidokat tartalmazó Gel Beads és az olaj egy mikrofluidikai chipen kombinálódnak, hogy az úgynevezett Gel Beads in Emulsion (GEM) reakcióvizikulákat alkossanak. A GEM-ek párhuzamosan képződnek a chip mikrofluidikai csatornáiban, lehetővé téve a felhasználó számára, hogy 100-10 000 egyedi sejtet dolgozzon fel egyetlen 7 perces Chromium™ műszerfutás során. Fontos megjegyezni, hogy a sejteket határhígításban töltik be annak érdekében, hogy maximalizálják az egyetlen sejtet tartalmazó GEM-ek számát az alacsony duplázódási arány biztosítása érdekében, miközben fenntartják a magas, akár ~65%-os sejtvisszanyerési arányt.
Minden funkcionális GEM egyetlen sejtet, egyetlen gélgyöngyöt és RT-reagenseket tartalmaz. Minden egyes GEM reakcióvázon belül egyetlen sejtet lizálnak, a Gel Bead-et feloldják, hogy az azonos vonalkódú RT oligonukleotidokat oldatba szabadítsák, és megtörténik a poliadenilált mRNS reverz transzkripciója. Ennek eredményeképpen az egyetlen sejtből származó összes cDNS azonos vonalkóddal rendelkezik, ami lehetővé teszi a szekvenálási leolvasások visszatérképezését az eredeti, egyetlen sejtből származó eredeti sejtekhez. Az NGS-könyvtárak előállítása ezekből a vonalkódolt cDNS-ekből ezután egy rendkívül hatékony tömeges reakcióban történik. Az alábbi videó nagyszerű vizuális magyarázatot ad arra, hogyan működik mindez.
A mintaelőkészítéstől a how-to videókig: A Chromium Single Cell 3′ munkafolyamat az érdeklődésre számot tartó sejtekkel kezdődik, majd ezt követi az NGS könyvtárépítés a reagens-készleteink és a Chromium Controller segítségével. A vonalkódolt cDNS könyvtár előkészítése (a fenti videóban általánosságban leírtak szerint) fontos lépés, és a “garbage in, garbage out” mondás szerint – a jó minőségű sejtszuszpenziók előkészítése kulcsfontosságú ahhoz, hogy a legtöbbet hozza ki az RNS-seq adatokból. Ha segítségre van szüksége a sejtelőkészítési folyamathoz, weboldalunkon több bemutatott protokoll is elérhető, mint például az idegszövetek disszociációja vagy a moha protoplaszt szuszpenziók előkészítése scRNA-seq használatához, és további protokollok rendszeresen kerülnek fel a támogatási weboldalunkra. Ha útmutatást és ajánlásokat szeretne kapni arra vonatkozóan, hogyan készítse elő legjobban a szuszpenzióban lévő mintáit, jó kiindulópont lehet az egysejtes előkészítési útmutató, ahol többet megtudhat a legjobb gyakorlatokról és megtekinthet egy általános protokollt. Ha inkább a vizuális tanulás híve, a how-to-videók, különösen a 4-7. fejezetek segítenek végigvezetni a minta- és könyvtár-előkészítés folyamatán.



-
Single Cell Prep Guide
-
Demonstrated Protocols
-
How…to-videók
-
Chromium™ Controller
-
User Guides
-
How-to-videók
-
Cell Ranger™ szoftver
-
Loupe™ Cell Browser
-
How-to-videók
Az adatok elemzése, sejtről sejtre
A könyvtár elkészítése után a szekvenálás Illumina® szekvenáló eszközökkel történik. Ez vezet a szórakoztató részhez: a biológiai meglátások kinyerése az egysejtes RNS-seq-adatokból! A Chromium Single Cell 3′ Solution által generált (potenciálisan) hatalmas adatmennyiség könnyen kezelhető szoftverünkkel és vizualizációs eszközeinkkel, amelyeket úgy terveztünk, hogy a találgatás nagy részét elvegyék – a Cell Ranger™ szoftver a vonalkódolt szekvenálási adatokat az egysejtes expressziós elemzésre kész fájlokká alakítja, a vizualizáció pedig a Loupe™ Cell Browser segítségével történik. Ha szeretne lépésről-lépésre útmutatót a szoftverünk használatáról, a hogyan-videóink 8-11. fejezete mindent tárgyal a Cell Ranger szoftverrel történő átírásszámlálástól a Loupe Cell Browserrel történő gyönyörű adatvizualizálásig. Egyre több, a közösség által kifejlesztett egysejtes elemző eszköz is létezik, köztük a Seurat, a Monocle és a Cell View. Olvasson többet ezekről az elemzőeszközökről és más egysejtes cikkekről a 10x Genomics Blogon, ahol kiemeljük a fontos kutatási hozzájárulásokat, tippeket és trükköket adunk, és naprakészen tájékoztatjuk Önt minden egysejtes dologról!
Ha bármilyen más kérdése vagy aggálya van a Chromium Single Cell 3′ Solution használatának megkezdésével kapcsolatban, nyugodtan hagyjon megjegyzést itt, vagy lépjen velünk közvetlenül kapcsolatba. Legyen szó az agyi organoidok fejlődésének nyomon követéséről, az akut myeloid leukémiában szenvedő betegek csontvelő-átültetésének tanulmányozásáról vagy a bélrendszeri őssejtek önmegújulásának új útvonalának feltárásáról, a Chromium Single Cell publikációk száma napról napra nő, és alig várjuk, hogy halljunk az Ön csodálatos kutatásairól!