Pływanie w morzu komórek: Why single-cell genomics?
Kompleksowe systemy biologiczne są wynikiem skoordynowanej funkcji poszczególnych komórek wykonujących skomplikowany „taniec”, z których każda wnosi do zespołu swoją własną, szczególną rolę. Ze względu na tę złożoność, badania ekspresji genów w organizmach, tkankach lub populacjach komórek są często ograniczone przez zastosowanie tradycyjnych metod masowego RNA-seq. Jako naukowcy, intuicyjnie rozumiemy, że kiedy „mielimy i znajdujemy” w celu przeprowadzenia eksperymentów na naszej rozcieńczonej mieszaninie RNA, tak naprawdę otrzymujemy tylko niewielki obraz tego, co może się dziać w interesujących nas komórkach: różnice międzykomórkowe są pomijane, a heterogeniczność komórek może być całkowicie zamaskowana. W rzeczywistości, masowa analiza ekspresji RNA często opisuje stan, w którym żadna (lub bardzo niewiele) z komórek w rzeczywistości nie istnieje!
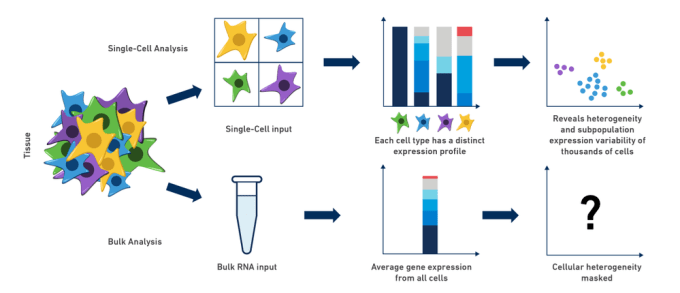
Prawdopodobnie jesteś neurobiologiem, pracującym w mózgu myszy, badającym rozwój subpopulacji neuronów, które produkują specyficzny neuroprzekaźnik. Gdybyś tylko mógł wyizolować tę populację i przeanalizować ją na zasadzie komórka po komórce, potencjalne spostrzeżenia byłyby rzeczywiście neuronalnie stymulujące! Podobnie w przypadku badań nad rakiem: jak najlepiej rozdzielić ekspresję genów i, co być może ważniejsze, heterogeniczność komórek w guzie? Co by się stało, gdyby można było porównać nie tylko różnice między guzami, ale także wewnątrz guza i zbadać populacje komórek odpornościowych związanych z mikrośrodowiskiem guza? Ta różnorodność, często przesłonięta przez metody zbiorczego RNA-seq, może być ujawniona przez użycie pojedynczych komórek RNA-seq do specyficznej eksploracji tych klinicznie istotnych populacji komórek. Przykłady wad analizy masowej są liczne, a analiza różnic między typami komórek w większości, jeśli nie we wszystkich, tych złożonych systemach biologicznych jest krytyczna dla naszego zrozumienia ich udziału, szczególnie podczas rozwoju i progresji choroby.
Jak izolujemy i analizujemy pojedyncze komórki?
Technologia single-cell RNA-seq (scRNA-seq) firmy 10x Genomics, Chromium™ Single Cell 3′ Solution, pozwala na analizę transkryptomów na zasadzie komórka po komórce poprzez zastosowanie mikroprzepływowego podziału w celu wychwycenia pojedynczych komórek i przygotowania barcode’owych bibliotek cDNA do sekwencjonowania następnej generacji (NGS). W szczególności, pojedyncze komórki, odczynniki do odwrotnej transkrypcji (RT), kulki żelowe zawierające zakodowane oligonukleotydy i olej są łączone na chipie mikroprzepływowym w celu utworzenia pęcherzyków reakcyjnych zwanych kulkami żelowymi w emulsji lub GEM. GEM-y są formowane równolegle w kanałach mikrofluidycznych chipa, co pozwala użytkownikowi na przetwarzanie od 100 do 10 000 pojedynczych komórek w ciągu jednego 7-minutowego przebiegu w urządzeniu Chromium™. Ważne jest, aby zauważyć, że komórki są ładowane w rozcieńczeniu ograniczającym, aby zmaksymalizować liczbę GEM-ów zawierających pojedynczą komórkę w celu zapewnienia niskiego wskaźnika dubletów, przy jednoczesnym utrzymaniu wysokiego wskaźnika odzysku komórek do ~65%.
Każdy funkcjonalny GEM zawiera pojedynczą komórkę, pojedynczą kulkę żelową oraz odczynniki RT. W każdym pęcherzyku reakcyjnym GEM, pojedyncza komórka jest lizowana, kulka żelowa jest rozpuszczana w celu uwolnienia identycznie zakodowanych oligonukleotydów RT do roztworu i następuje odwrotna transkrypcja poliadenylowanego mRNA. W rezultacie wszystkie cDNA z pojedynczej komórki będą miały ten sam kod kreskowy, co pozwala na mapowanie odczytów sekwencjonowania z powrotem do ich oryginalnych pojedynczych komórek, z których pochodzą. Przygotowanie bibliotek NGS z tych zakodowanych cDNA jest następnie przeprowadzane w wysoce wydajnej reakcji masowej. Poniższy film wideo doskonale wyjaśnia, jak to wszystko działa.
Od przygotowania próbki do filmów how-to: Pomocne linki do rozpoczęcia pracy
Przebieg pracy Chromium Single Cell 3′ rozpoczyna się od przygotowania interesujących Państwa komórek, po czym następuje konstrukcja biblioteki NGS przy użyciu naszych zestawów odczynników i urządzenia Chromium Controller. Przygotowanie biblioteki cDNA z kodem paskowym (jak ogólnie opisano w powyższym filmie) jest ważnym krokiem i zgodnie z powiedzeniem „garbage in, garbage out” – przygotowanie wysokiej jakości zawiesin komórkowych jest kluczem do uzyskania jak największej ilości danych RNA-seq. Jeśli potrzebujesz pomocy w procesie przygotowania komórek, mamy wiele zademonstrowanych protokołów dostępnych na naszej stronie internetowej, takich jak dysocjacja tkanki nerwowej lub przygotowanie zawiesiny protoplastów mchu do użycia w scRNA-seq, a kolejne protokoły są regularnie dodawane na naszej stronie wsparcia. Aby uzyskać wskazówki i zalecenia dotyczące najlepszego sposobu przygotowania próbek w zawiesinie, dobrym punktem wyjścia jest nasz przewodnik przygotowania pojedynczych komórek, gdzie można dowiedzieć się więcej o najlepszych praktykach i obejrzeć ogólny protokół. Jeśli jesteś bardziej wzrokowcem, nasze filmy, szczególnie rozdziały od 4 do 7, powinny pomóc Ci przeprowadzić Cię przez proces przygotowania próbek i bibliotek.



-
Przewodnik przygotowania pojedynczej komórki
-
Demonstrowane protokoły
-
How-to-videos
-
Chromium™ Controller
-
User Guides
-
How-to-videos
-
Cell Ranger™ Software
-
Loupe™ Cell Browser
-
How-to-videos
Analiza Twoich danych, jedna komórka na raz
Po przygotowaniu biblioteki wykonywane jest sekwencjonowanie przy użyciu instrumentów do sekwencjonowania Illumina®. To prowadzi do najciekawszej części: wyciągnięcia wniosków biologicznych z danych RNA-seq z pojedynczej komórki! (Potencjalnie) ogromna ilość danych generowanych przez Chromium Single Cell 3′ Solution może być z łatwością obsłużona przez nasze oprogramowanie i narzędzia wizualizacyjne, które zostały zaprojektowane w celu wyeliminowania wielu zgadywanek – oprogramowanie Cell Ranger™ przekształca dane sekwencjonowania z kodem kreskowym w pliki gotowe do analizy ekspresji pojedynczych komórek, a wizualizacja jest wykonywana za pomocą przeglądarki Loupe™ Cell Browser. Jeśli chcesz poznać krok po kroku sposób korzystania z naszego oprogramowania, rozdziały od 8 do 11 naszych filmów instruktażowych obejmują wszystko, od liczenia transkryptów za pomocą oprogramowania Cell Ranger Software do pięknej wizualizacji danych za pomocą przeglądarki Loupe Cell Browser. Istnieje również rosnąca liczba narzędzi do analizy pojedynczych komórek, opracowanych przez społeczność, w tym Seurat, Monocle i Cell View. Przeczytaj więcej o tych narzędziach analitycznych i innych artykułach dotyczących pojedynczych komórek na blogu 10x Genomics Blog, gdzie podkreślamy ważny wkład w badania, dostarczamy wskazówek i sztuczek oraz informujemy na bieżąco o wszystkich rzeczach związanych z pojedynczymi komórkami!
Jeśli masz jakiekolwiek inne pytania lub wątpliwości dotyczące rozpoczęcia pracy z Chromium Single Cell 3′ Solution, możesz zostawić komentarz tutaj lub skontaktować się z nami bezpośrednio. Niezależnie od tego, czy chodzi o śledzenie rozwoju organoidów mózgu, badanie przeszczepu szpiku kostnego u pacjentów z ostrą białaczką szpikową, czy też odkrycie nowej ścieżki samoodnowy jelitowych komórek macierzystych, liczba publikacji Chromium Single Cell rośnie z dnia na dzień, a my z niecierpliwością czekamy, aby usłyszeć o wszystkich Twoich niesamowitych badaniach!