Svømning i et hav af celler:
Komplekse biologiske systemer er resultatet af den koordinerede funktion af individuelle celler, der udfører en indviklet “dans”, hvor hver enkelt celle bidrager med sin egen særlige rolle til helheden. På grund af denne kompleksitet er forskning i genekspression i organismer, væv eller cellepopulationer ofte begrænset ved at anvende traditionelle bulk RNA-seq-metoder. Som videnskabsmænd forstår vi intuitivt, at når vi “sliber og finder” for at udføre eksperimenter på vores fortyndede blanding af RNA, får vi i virkeligheden kun en lille idé om, hvad der kan foregå i vores celler af interesse: forskelle fra celle til celle overses, og cellulær heterogenitet kan blive fuldstændig maskeret. Faktisk beskriver bulk-RNA-ekspressionsanalyser ofte en formodet tilstand, hvor ingen (eller meget få) af cellerne faktisk eksisterer!
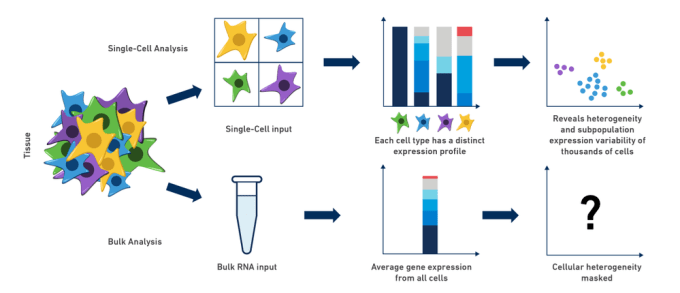
Måske er du en neurovidenskabsmand, der arbejder i musens hjerne og studerer udviklingen af en subpopulation af neuroner, der producerer en bestemt neurotransmitter. Hvis du blot kunne isolere denne population og analysere den celle for celle, ville den potentielle indsigt virkelig være neuronalt stimulerende! Tilsvarende gælder for kræftforskning: Hvordan kan man bedst skille genudtryk og, måske endnu vigtigere, cellulær heterogenitet i en tumor ad? Hvad hvis man kunne sammenligne ikke kun forskelle mellem tumorer, men også inden for tumorerne og undersøge de immuncellepopulationer, der er relateret til tumormikromiljøet? Denne mangfoldighed, der ofte er skjult af bulk RNA-seq-metoder, kan afsløres ved at bruge enkeltcellers RNA-seq til specifikt at udforske disse klinisk betydningsfulde cellepopulationer. Eksemplerne på ulemper ved bulkanalyse er rigelige, og dissekering af celletypeforskelle i de fleste, om ikke alle, af disse komplekse biologiske systemer er afgørende for vores forståelse af deres bidrag, især under udvikling og i sygdomsforløbet.
Hvordan isolerer og analyserer vi enkeltceller?
10x Genomics’ single-cell RNA-seq (scRNA-seq)-teknologi, Chromium™ Single Cell 3′ Solution, giver dig mulighed for at analysere transkriptomer celle for celle ved hjælp af mikrofluidisk partitionering til at indfange enkelte celler og forberede stregkodede, næste-generations sekventering (NGS) cDNA-biblioteker. Specifikt kombineres enkeltceller, omvendt transskription (RT) reagenser, Gel Beads indeholdende stregkodede oligonukleotider og olie på en mikrofluidisk chip for at danne reaktionsvesikler kaldet Gel Beads in Emulsion, eller GEMs. GEM’er dannes parallelt i de mikrofluidiske kanaler i chippen, hvilket giver brugeren mulighed for at behandle 100 til 10.000 enkeltceller i en enkelt 7-minutters Chromium™ Instrument-kørsel. Det er vigtigt at bemærke, at cellerne belastes ved en begrænsende fortynding for at maksimere antallet af GEM’er, der indeholder en enkelt celle, for at sikre en lav doubletrate, samtidig med at der opretholdes en høj cellegenvindingsrate på op til ~65%.
Hver funktionel GEM indeholder en enkelt celle, en enkelt Gel Bead og RT-reagenser. I hver GEM-reaktionsvesikel lyseres en enkelt celle, Gel Bead opløses for at frigøre de identisk stregkodede RT-oligonukleotider i opløsningen, og der sker en omvendt transskription af polyadenyleret mRNA. Som et resultat heraf vil alle cDNA’er fra en enkelt celle have den samme stregkode, hvilket gør det muligt at kortlægge sekventeringslæsningerne tilbage til deres oprindelige oprindelsescelle. Udarbejdelsen af NGS-biblioteker fra disse stregkodede cDNA’er udføres derefter i en meget effektiv bulkreaktion. Videoen nedenfor giver en god visuel forklaring på, hvordan det hele fungerer.
Fra prøveforberedelse til vejledningsvideoer: Nyttige links til at komme i gang
The Chromium Single Cell 3′ workflow begynder med dine celler af interesse efterfulgt af NGS-bibliotekskonstruktion ved hjælp af vores reagenssæt og Chromium Controller. Forberedelse af dit stregkodede cDNA-bibliotek (som generelt beskrevet i ovenstående video) er et vigtigt skridt, og tro mod ordsproget “garbage in, garbage out” – forberedelse af cellesuspensioner af høj kvalitet er nøglen til at få mest muligt ud af dine RNA-seq-data. Hvis du har brug for hjælp til celleforberedelsesprocessen, har vi flere demonstrerede protokoller tilgængelige på vores websted, f.eks. dissociering af neuralt væv eller forberedelse af mosprotoplastsuspensioner til brug i scRNA-seq, og flere protokoller tilføjes regelmæssigt på vores supportwebsted. For vejledning og anbefalinger om, hvordan du bedst forbereder dine prøver, når de er i suspension, er vores guide til forberedelse af enkeltceller et godt udgangspunkt, hvor du kan få mere at vide om bedste praksis og se en generel protokol. Hvis du er mere til visuel læring, kan vores hvordan-til-videoer, især kapitel 4 til 7, hjælpe dig med at gå gennem prøve- og biblioteksforberedelsesprocessen.



-
Single Cell Prep Guide
-
Demonstrerede protokoller
-
How-to-videoer
-
Chromium™ Controller
-
Brugervejledninger
-
Hvordan-to-videoer
-
Cell Ranger™ Software
-
Loupe™ Cell Browser
-
How-to-videos
Analyse af dine data, en celle ad gangen
Når dit bibliotek er forberedt, udføres sekventering ved hjælp af Illumina®-sekventeringsinstrumenter. Dette fører til den sjove del: at uddrage biologiske indsigter fra dine RNA-seq-data fra enkeltceller! Den (potentielt) enorme mængde data, som du genererer fra Chromium Single Cell 3′-løsningen, kan nemt håndteres af vores software og visualiseringsværktøjer, der er designet til at fjerne en stor del af gætteriet – Cell Ranger™ Software omdanner de stregkodede sekventeringsdata til filer, der er klar til enkeltcelleekspressionsanalyse, og visualiseringen sker via Loupe™ Cell Browser. Hvis du gerne vil have en trinvis vejledning i brugen af vores software, dækker kapitel 8 til 11 i vores how-to-videos alt fra transkripttælling med Cell Ranger Software til smuk datavisualisering med Loupe Cell Browser. Der er også et voksende antal enkeltcelleanalyseværktøjer, der er udviklet af fællesskabet, herunder Seurat, Monocle og Cell View. Læs mere om disse analyseværktøjer og andre artikler om enkeltceller på 10x Genomics Blog, hvor vi fremhæver vigtige forskningsbidrag, giver tips og tricks og holder dig opdateret om alt, hvad der vedrører enkeltceller!
Hvis du har andre spørgsmål eller bekymringer om at komme i gang med Chromium Single Cell 3′ Solution, er du velkommen til at efterlade en kommentar her eller kontakte os direkte. Uanset om det drejer sig om at spore hjernens organoidudvikling, studere knoglemarvstransplantation hos patienter med akut myeloid leukæmi eller afdække en ny vej for tarmens stamcelle-selvfornyelse, vokser antallet af Chromium Single Cell-publikationer dagligt, og vi ser frem til at høre om al din fantastiske forskning!