Simmar i ett hav av celler:
Komplexa biologiska system är resultatet av den samordnade funktionen hos enskilda celler som utför en invecklad ”dans”, där var och en bidrar med sin egen speciella roll till helheten. På grund av denna komplexitet är forskning om genuttryck i organismer, vävnader eller cellpopulationer ofta begränsad genom att använda traditionella bulk RNA-seq-metoder. Som forskare förstår vi intuitivt att när vi ”slipar och letar” för att utföra experiment på vår utspädda blandning av RNA, får vi egentligen bara en liten bild av vad som kan hända i de celler vi är intresserade av. Skillnader mellan cellerna missas och den cellulära heterogeniteten kan maskeras helt och hållet. Faktum är att analysen av RNA-uttryck i bulk ofta beskriver ett tillstånd i vilket ingen (eller mycket få) av cellerna faktiskt existerar!
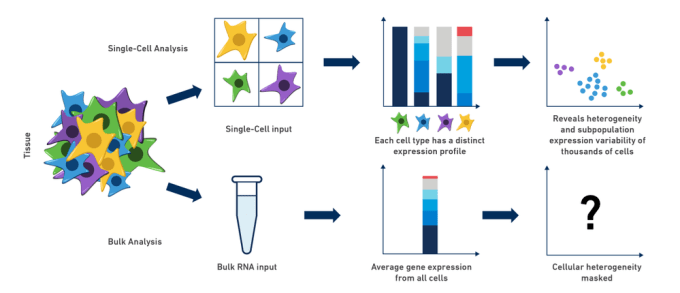
Kanske är du en neurovetenskapsman som arbetar i mushjärnan och studerar utvecklingen av en subpopulation av neuroner som producerar en specifik neurotransmittor. Om du bara kunde isolera den populationen och analysera den cell för cell skulle de potentiella insikterna verkligen vara neuronalt stimulerande! På samma sätt är det när det gäller cancerforskning: hur kan man bäst skilja ut genuttryck och, kanske ännu viktigare, cellulär heterogenitet i en tumör? Tänk om man kunde jämföra skillnader inte bara mellan tumörer utan även inom tumörer och utforska de immuncellspopulationer som är relaterade till tumörens mikromiljö? Denna mångfald, som ofta döljs av bulk RNA-seq-metoder, kan avslöjas genom att använda RNA-seq för enskilda celler för att specifikt utforska dessa kliniskt betydelsefulla cellpopulationer. Exemplen på nackdelar med bulkanalyser är många, och att dissekera skillnader mellan celltyper i de flesta, om inte alla, av dessa komplexa biologiska system är avgörande för vår förståelse av deras bidrag, särskilt under utveckling och vid sjukdomsutveckling.
Hur isolerar och analyserar vi enskilda celler?
10x Genomics RNA-seq-teknik för enskilda celler (scRNA-seq), Chromium™ Single Cell 3′ Solution, gör det möjligt att analysera transkriptom på cell-för-cell-basis genom användning av mikrofluidisk partitionering för att fånga upp enskilda celler och förbereda streckkodade, nästa generations sekvensering (NGS) cDNA-bibliotek. Specifikt kombineras enskilda celler, reagens för omvänd transkription (RT), Gel Beads som innehåller streckkodade oligonukleotider och olja på ett mikrofluidiskt chip för att bilda reaktionsvesiklar som kallas Gel Beads in Emulsion, eller GEMs. GEMs bildas parallellt i chipets mikrofluidiska kanaler, vilket gör det möjligt för användaren att bearbeta 100 till 10 000 enskilda celler i en enda 7-minuters körning med Chromium™ Instrument. Det är viktigt att notera att cellerna laddas vid en begränsande utspädning för att maximera antalet GEMs som innehåller en enskild cell för att säkerställa en låg dubblettfrekvens, samtidigt som en hög cellåtervinningsfrekvens på upp till ~65 % bibehålls.
Varje funktionell GEM innehåller en enskild cell, en enskild Gel Bead och RT-reagenser. I varje GEM-reaktionsvesikel lyseras en enda cell, Gel Bead löses upp för att frigöra de identiskt streckkodade RT-oligonukleotiderna i lösningen, och omvänd transkription av polyadenylerat mRNA sker. Som ett resultat av detta kommer alla cDNA från en enskild cell att ha samma streckkod, vilket gör det möjligt att kartlägga sekvenseringsläsningarna tillbaka till deras ursprungliga enskilda ursprungsceller. Beredningen av NGS-bibliotek från dessa streckkodade cDNA:er utförs sedan i en mycket effektiv bulkreaktion. Videon nedan ger en bra visuell förklaring av hur allt detta fungerar.
Från provberedning till instruktionsvideor: Chromium Single Cell 3′-arbetsflödet börjar med dina celler av intresse följt av NGS-bibliotekskonstruktion med hjälp av våra reagenskit och Chromium Controller. Att förbereda ditt streckkodade cDNA-bibliotek (enligt den allmänna beskrivningen i videon ovan) är ett viktigt steg och enligt ordspråket ”garbage in, garbage out” – att förbereda högkvalitativa cellsuspensioner är nyckeln till att få ut det mesta av dina RNA-seq-data. Om du behöver hjälp med cellberedningsprocessen har vi flera demonstrerade protokoll tillgängliga på vår webbplats, t.ex. dissociering av neurala vävnader eller beredning av mossprotoplastsuspensioner för användning i scRNA-seq, och fler protokoll läggs regelbundet till på vår supportwebbplats. För vägledning och rekommendationer om hur du bäst förbereder dina prover när de väl är suspenderade är en bra utgångspunkt vår guide för beredning av enskilda celler, där du kan lära dig mer om bästa praxis och se ett allmänt protokoll. Om du är mer av en visuell inlärare bör våra instruktionsvideor, särskilt kapitel 4 till 7, hjälpa dig att gå igenom prov- och biblioteksberedningsprocessen.



-
Single Cell Prep Guide
-
Demonstrerade protokoll
-
How-videor
-
Chromium™ Controller
-
Användarhandböcker
-
Hur-to-videos
-
Cell Ranger™ Software
-
Loupe™ Cell Browser
-
How-to-videos
Analysera dina data, en cell i taget
När ditt bibliotek har förberetts utförs sekvensering med Illumina®-sekvenseringsinstrument. Detta leder till den roliga delen: att hämta biologiska insikter från dina RNA-seq-data från en enskild cell! Den (potentiellt) enorma mängden data som du genererar från Chromium Single Cell 3′-lösningen kan enkelt hanteras av vår programvara och våra visualiseringsverktyg som utformats för att ta bort en stor del av gissningsarbetet – Cell Ranger™ Software omvandlar de streckkodade sekvenseringsdata till filer som är färdiga för uttrycksanalys av enstaka celler, och visualiseringen sker via Loupe™ Cell Browser. Om du vill ha en steg-för-steg-handledning om hur du använder vår programvara täcker kapitel 8 till 11 i våra instruktionsvideor allt från transkripträkning med Cell Ranger Software till vacker datavisualisering med Loupe Cell Browser. Det finns också ett växande antal analysverktyg för enskilda celler som utvecklats av gemenskapen, inklusive Seurat, Monocle och Cell View. Läs mer om dessa analysverktyg och andra artiklar om enskilda celler på 10x Genomics Blog där vi lyfter fram viktiga forskningsbidrag, ger tips och tricks och håller dig uppdaterad om allt som rör enskilda celler!
Om du har några andra frågor eller funderingar om hur du kommer igång med Chromium Single Cell 3′-lösningen är du välkommen att lämna en kommentar här eller kontakta oss direkt. Vare sig det handlar om att spåra hjärnans organoidutveckling, studera benmärgstransplantation hos patienter med akut myeloisk leukemi eller avslöja en ny väg för självförnyelse av tarmstamceller, så ökar antalet Chromium Single Cell-publikationer dagligen, och vi ser fram emot att få höra talas om all din fantastiska forskning!