COSMIC, the Catalogue Of Somatic Mutations In Cancer, prowadzony przez Wellcome Trust Sanger Institute, jest jednym z największych i najbardziej wszechstronnych zasobów do badania wpływu mutacji somatycznych w ludzkim raku. Jest to produkt ogromnego wysiłku ekspertów w zakresie kurateli danych, prezentujący dane z tysięcy publikacji i wielu ważnych zestawów danych genomiki nowotworów o dużej skali. COSMIC działa jako główny portal dla co najmniej czterech ważnych projektów lub zasobów: COSMIC – the Catalogue Of Somatic Mutations In Cancer, the Cancer Gene Census, the Cell Lines Project oraz COSMIC-3D. Użytkownicy akademiccy mogą używać i pobierać (po zarejestrowaniu) dane za darmo, o ile nie będą ich ponownie rozpowszechniać i zgodzą się na warunki licencji. Użytkownicy nastawieni na zysk muszą wnieść opłatę, aby pobrać COSMIC. Te zasoby zapewniają dostęp do bardzo bogatego zestawu danych, wizualizacji i interpretacji dla zrozumienia mutacji nowotworowych i genów nowotworowych.
Baza danych COSMIC
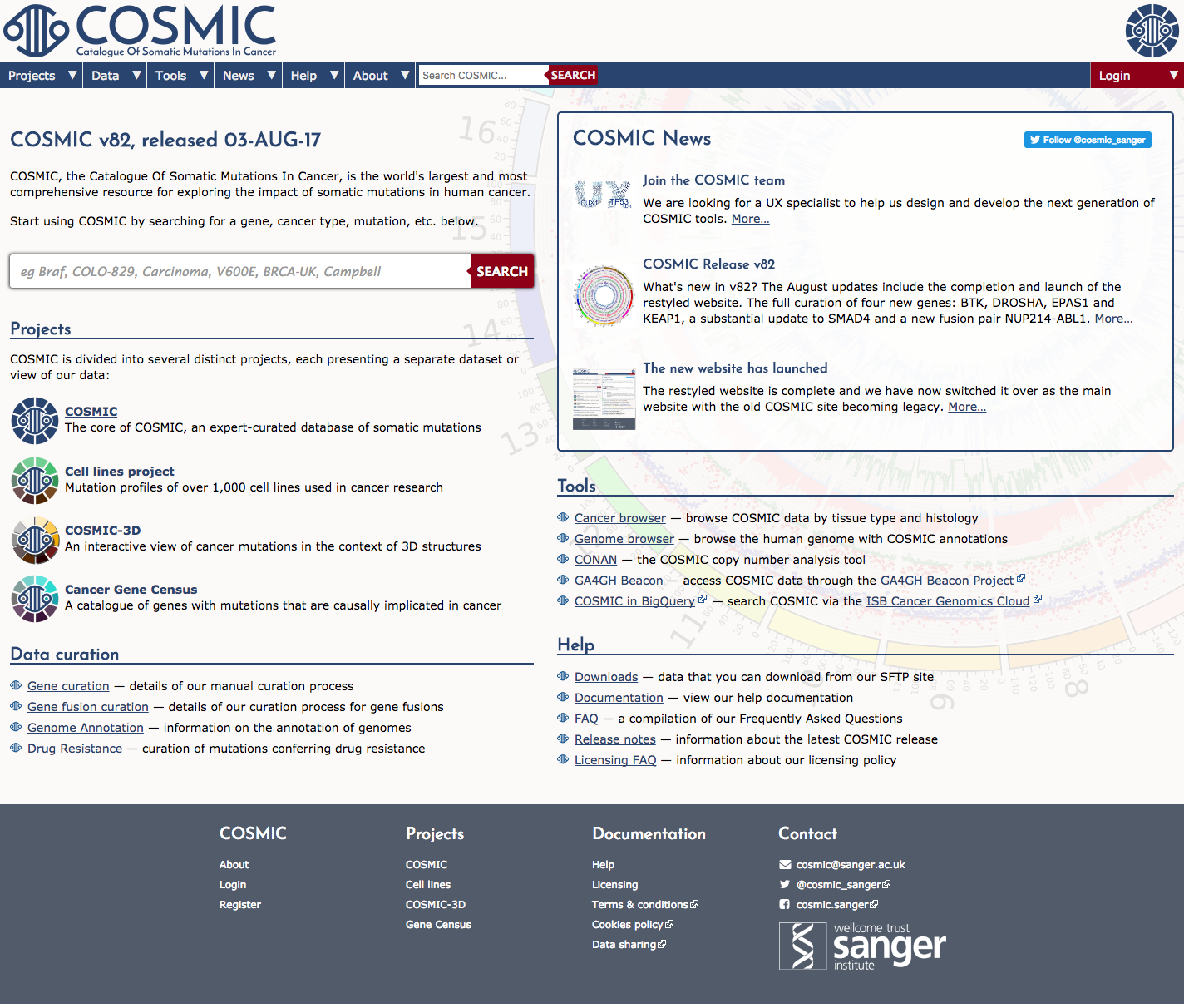
Aby przeglądać COSMIC możesz po prostu przejść do strony głównej i wyszukać gen, typ nowotworu, mutację, itp. w polu wyszukiwania. Dla ilustracji przeanalizujemy wyniki dla pojedynczego genu. Wpisz BRAF w interfejsie wyszukiwania i naciśnij enter. W czasie pisania tego tekstu, to hasło wyszukiwania zwróciło wyniki dla 1 genu, 826 mutacji, 49 nowotworów, 226 próbek, 1575 cytowań Pubmed i 0 badań (zobacz zakładki wyników). Należy zauważyć, że COSMIC zawiera wyniki dla prawie 300 000 przebadanych próbek i ponad 50 000 mutacji w BRAF.
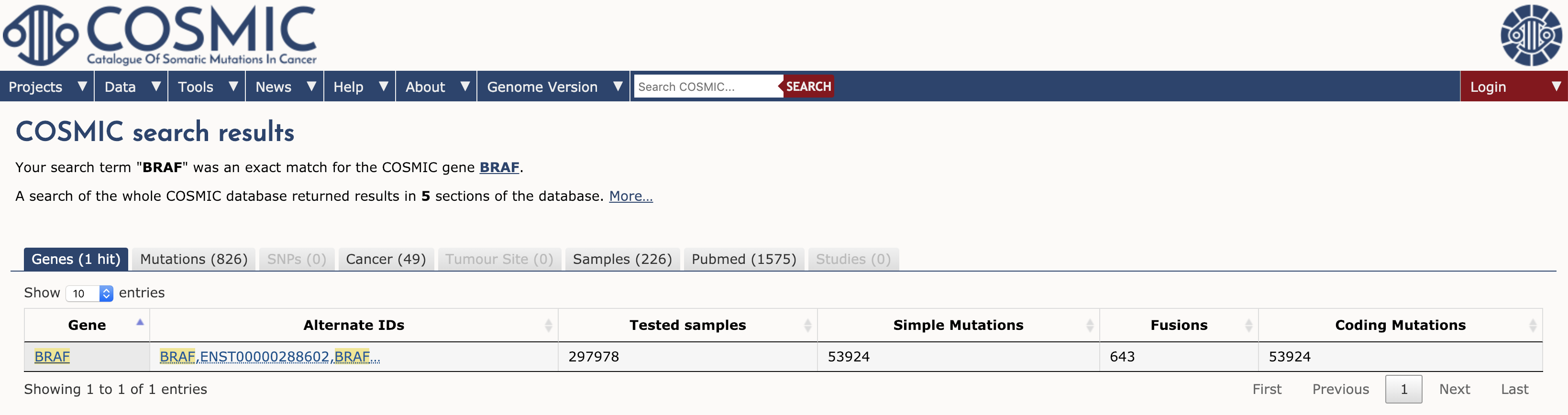
Jeśli wybierzemy wynik BRAF, COSMIC zwraca szczegółową stronę, która zawiera: streszczenia genów, linki do innych zasobów COSMIC (np. geny Census, geny Hallmark, itp.), linki zewnętrzne, oporność na leki, dystrybucję tkanek, widok przeglądarki genomu, dystrybucję mutacji, warianty i referencje. Przyjrzymy się kilku z tych sekcji. Po pierwsze, spójrzmy na sekcję Overview. Wzdłuż górnej części tej sekcji znajduje się kilka użytecznych ikon. Ikona „gen ze spisu” mówi nam, że BRAF jest znanym genem nowotworowym zgodnie ze spisem genów (patrz poniżej). Kolejne trzy ikony informują nas, że jest to również „Gen nadzorowany przez ekspertów”, że eksperymenty mutagenezy insercyjnej na myszach potwierdzają, że BRAF jest genem nowotworowym, i wreszcie, że BRAF jest genem „Cancer Hallmark”. Po tych ikonach jest wiele więcej szczegółów o BRAF, w tym współrzędne, synonimy, link do COSMIC-3D (patrz poniżej), i więcej.
Następnie zbadajmy widok genu. Histogram częstotliwości mutacji (substytucji) pokazuje bardzo dramatyczny „hotspot” mutacji w pozycji 600 (np., p.V600E). Najedź myszką na tę część histogramu, aby zobaczyć szczegóły. Jest to bardzo dobrze znana mutacja sterująca w wielu typach nowotworów.
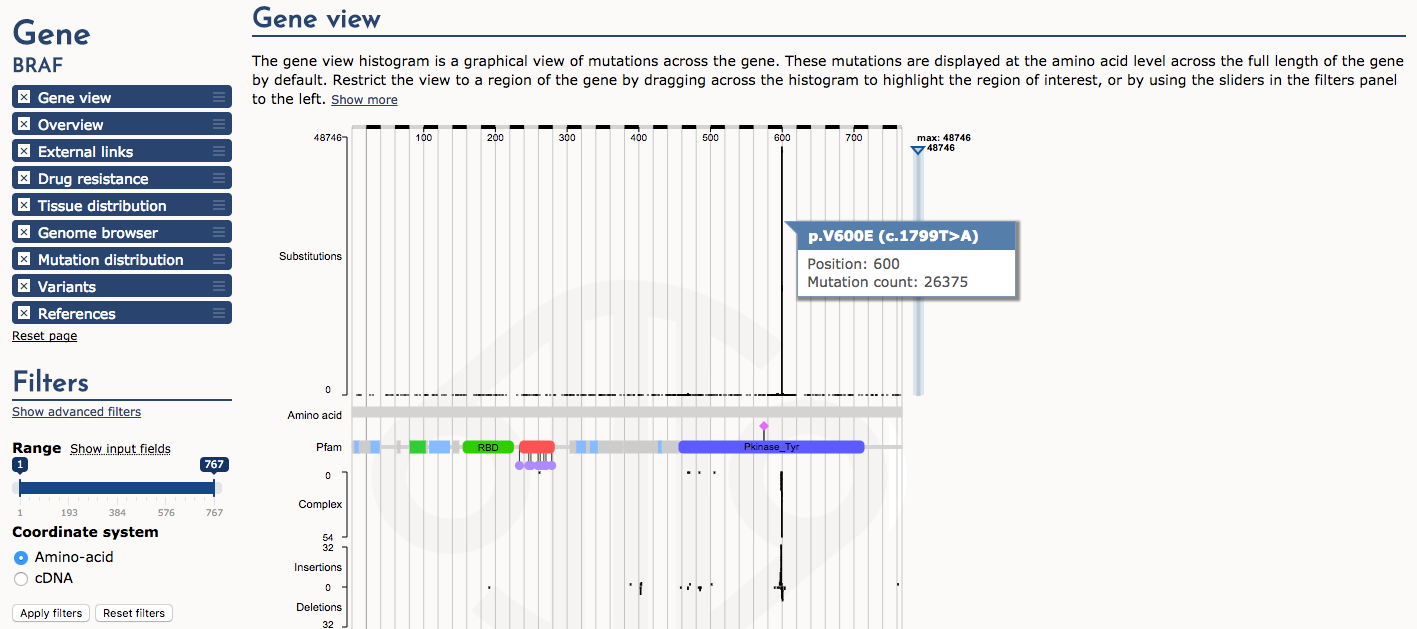
Jakiej części (domeny) białka BRAF dotyczy mutacja p.V600E?
Domena kinazy tyrozynowej białka (Pfam)
Na koniec przejdź do sekcji „Dystrybucja tkankowa”. Posortuj tabelę według „mutacji punktowych” -> „% Mutated”. Zauważ, że nowotwory tarczycy i skóry (np. czerniak) są zdecydowanie najbardziej konsekwentnie zmutowane w locus genu BRAF (uwaga: NS oznacza nieokreślony). Pewna podgrupa próbek wykazuje również zmiany liczby kopii (CNV) i podwyższoną ekspresję. Ogólnie rzecz biorąc, niektóre przeważnie zmutowane geny mają tendencję do wiązania się z nowotworami określonego pochodzenia. Istnieje jednak wiele wyjątków od tego stwierdzenia i niektóre geny (np, TP53) są szeroko zmutowane w wielu różnych typach nowotworów.
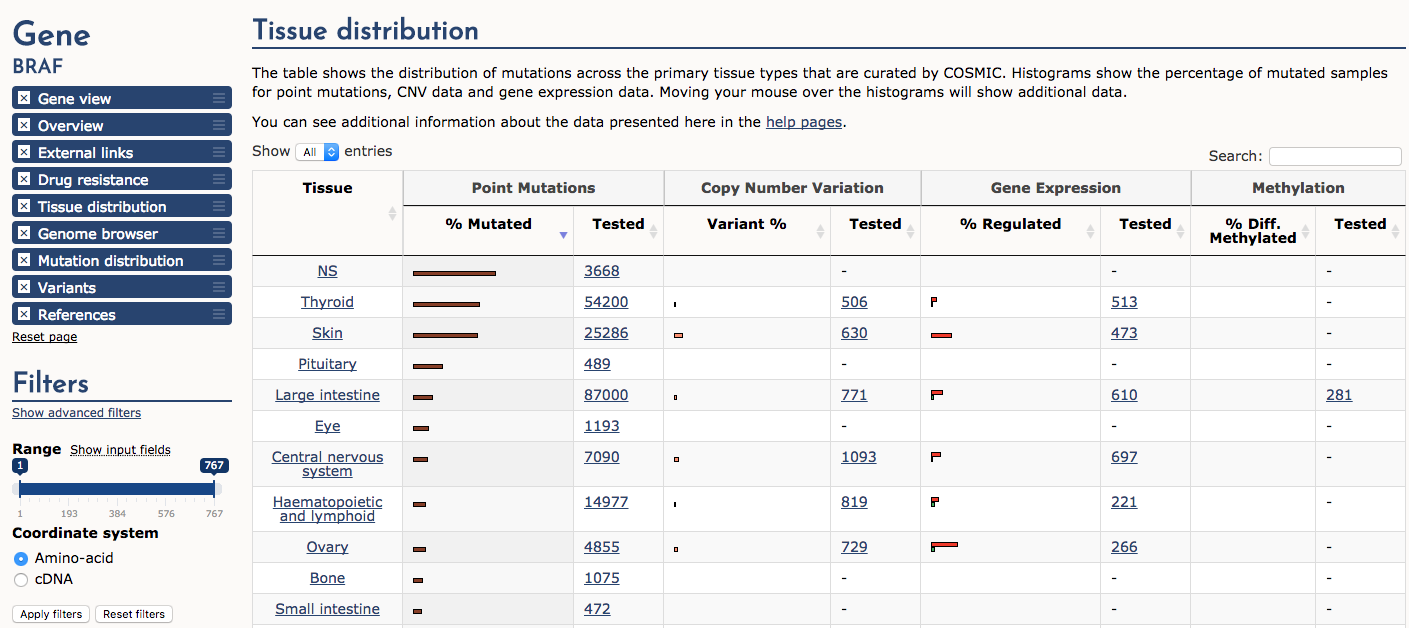
Jaki typ tkanki nowotworowej jest najczęściej dotknięty nadekspresją BRAF?
Około 15% raków jajnika jest dotkniętych nadekspresją BRAF
Cancer Gene Census
Cancer Gene Census (CGC) jest ciągłym wysiłkiem mającym na celu skatalogowanie tych genów, dla których mutacje (somatyczne lub germinalne) zostały przyczynowo powiązane z rakiem. Oryginalny spis i analiza została opublikowana w Nature Reviews Cancer przez Futreal et al. 2004, ale nadal otrzymuje regularne aktualizacje. CGC jest powszechnie uważany za ostateczną listę genów nowotworowych (supresorów nowotworów i onkogenów). Przejdź do Cancer Gene Census z rozwijanego menu 'Projects’ dostępnego na każdej stronie COSMIC. Strona ta jest podzielona na trzy główne sekcje: Cancer Gene Census, Breakdown oraz Abbreviations. W pierwszej sekcji wyświetlana jest prosta tabela wszystkich genów Cancer Gene Census.
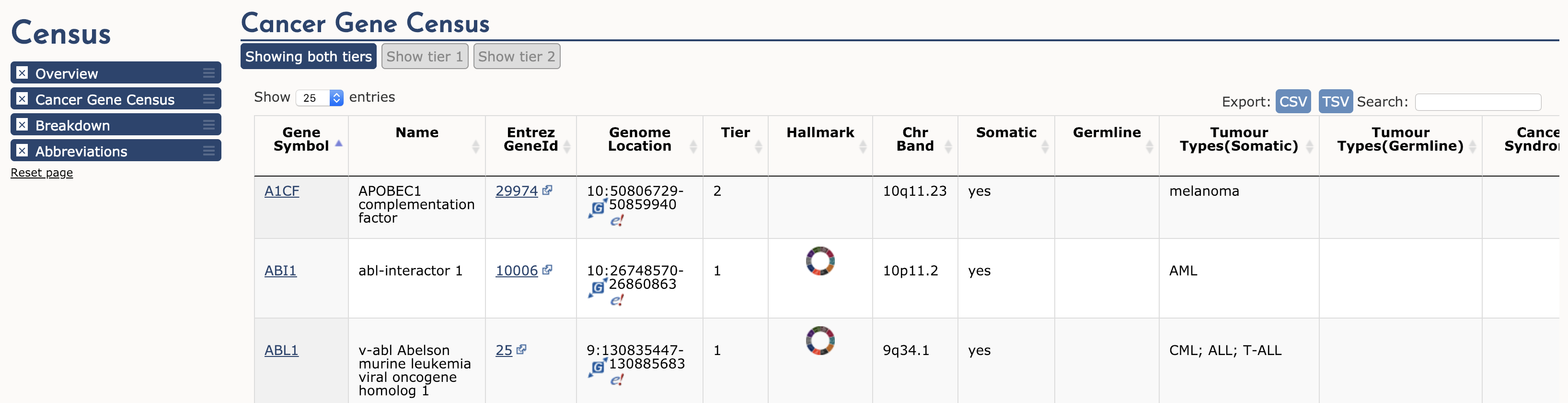
Aby to zilustrować, zbadajmy ABL1 (trzeci gen w tabeli). Pierwsze cztery kolumny zawierają nazwę opisową genu, linki do jego stron COSMIC i Entrez gene oraz region genomowy z linkami do widoków COSMIC i Ensembl Browser. Pozostałe kolumny informują nas, że ABL1 jest zlokalizowany na chromosomie 9q34.1, o którym wiadomo, że jest somatycznie zmutowany w CML, ALL i T-ALL. Działa on jako onkogen i partner do fuzji genów. W rzeczywistości ABL1 jest częścią prawdopodobnie najbardziej znanej fuzji nowotworowej, BCR-ABL, produktu rearanżacji chromosomu Philadelphia. Ta fuzja definiuje i napędza prawie wszystkie przypadki CML, a jej odkrycie doprowadziło do jednego z najbardziej udanych przykładów terapii celowanej (Imatinib). Aby dowiedzieć się więcej na temat roli ABL1 w raku, wybierz ikonę 'Census Hallmark’. Hallmark” jest odniesieniem do przełomowej pracy Hanahan i Weinberg (2000), zaktualizowanej w 2011 roku, w której zaproponowali oni, że wszystkie nowotwory mają sześć (obecnie dziesięć) wspólnych cech (hallmarks), które wyjaśniają transformację normalnych komórek w złośliwe komórki nowotworowe. Kuratorzy Cancer Gene Census starali się przypisać każdy gen nowotworowy w spisie do jednego lub więcej z tych cech. Grafika pokazuje, które z tych cech są promowane (zielone paski) lub tłumione (niebieskie paski) przez ABL1. W tym przypadku uważa się, że ABL1 promuje sygnalizację proliferacyjną, zmianę energetyki komórkowej, niestabilność genomu, angiogenezę, inwazję i przerzuty oraz tłumi programowaną śmierć komórki.
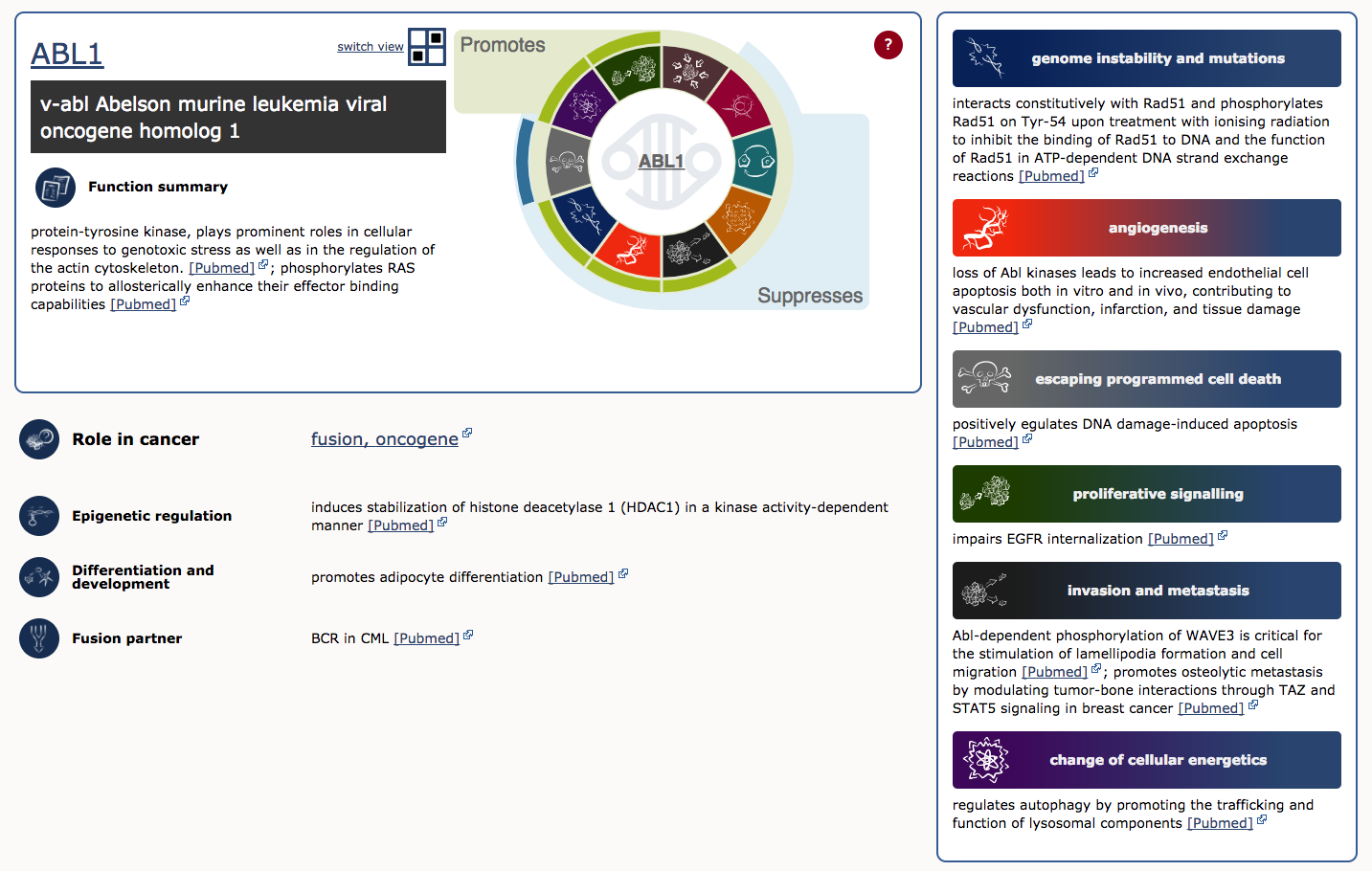
Ile genów supresorowych nowotworów (TSGs) i onkogenów znajduje się w CGC?
W czasie pisania tekstu w CGC znajdowało się 315 TSGs i 315 onkogenów. Co ciekawe, istnieją 72 geny, które funkcjonują zarówno jako onkogeny jak i TSG.
Uwaga: Musisz zarejestrować się w COSMIC, aby pobrać kompletny Cancer Gene Census. Następnie możesz załadować ten plik do R i użyć linuxowego wiersza poleceń lub innego podejścia, aby określić listę TSG i onkogenów obecnie udokumentowanych w CGC.
.